Haplogruppe R (Y-DNA)
- Wikipedia:Vorlagenfehler/Vorlage:Cite journal/temporär
- Haplogruppe
- Wikipedia:Vorlagenfehler/Vorlage:Cite book/temporär
| Haplogruppe des Y-Chromosoms | |
|---|---|
| Name | R |
| Ursprungsdatum | vor 26.800 Jahren |
| Ursprungsort | Zentralasien oder Südasien |
| Vorfahr | P |
| Nachkommen | R1, R1a, R1b, R2 |
| Mutationen |
|
Y-Haplogruppe R ist in der Humangenetik eine Haplogruppe des Y-Chromosoms. Sie ist eine Untergruppe von P. Die Mutation M207 trägt zu ihrer Unterscheidung bei.
Ursprung
Es wird angenommen, dass die Haplogruppe R in Nordwestasien vor 30.000 bis 35.000 Jahren erstmals auftrat. Die eher seltenen Formen der Chromosomen der Haplogruppe R, sowie die häufigsten Fälle der mit ihr eng verwandten Haplogruppe Q, werden interessanterweise unter Bevölkerungen in Mittelasien, in Südasien, in Australien, in Sibirien, bei amerikanischen Ureinwohnern, in Ägypten und in Kamerun gefunden. Sie ist die häufigste Haplogruppe in Europa.
Kivisild et al. schlagen vor, dass Süd- und Westasien der Ursprung dieser Haplogruppe" sein könnte:
Anhand der geographischen Verbreitung und der STR Unterschiede der Schwesterngruppen R1 and R2 (letztere ist auf Indien, Pakistan, Iran und das südliche Zentralasien beschränkt) ist es möglich dass Süd- und Westasien die Quelle für die Abgrenzung von R1 und R2 sind.
Verbreitung
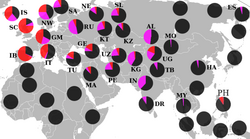
Die Mehrheit der Träger von Haplogruppe R gehören der Haplogruppe R1 an, die durch Marker M173 unterschieden wird. R1 ist in Europa und Westeurasien sehr verbreitet. Es wird angenommen, dass seine Verbreitung mit der Wiederbesiedelung von Nord-Eurasien nach der letzten Eiszeit zusammenhängt. Seine Hauptuntergruppen sind R1a (SRY1532) und R1b (M343). Ein isolierter Stamm der Y-Chromosomen, die scheinbar zu Haplogruppe R1b1* (P25) gehören, wurde in hoher Konzentration unter der einheimischen Bevölkerung von Nordkamerun in West-Zentralafrika gefunden. Es wird angenommen, dass dies eine prähistorische Rückwanderung einer alten protoeurasischen Bevölkerung nach Afrika darstellt. Einige Forscher haben auch über Haplogruppe T Y-Chromosomen in geringer Konzentration in einigen dieser kamerunischen Bevölkerungen berichtet, die eine eurasische Ähnlichkeit aufweisen. [1] Einige Y-Chromosomen, die scheinbar mit den kamerunischen R1b1* Chromosomen eng verwandt sind, werden in hoher Konzentration unter der modernen Bevölkerung in Ägypten gefunden. Viele moderne Bevölkerungen des Nordkamerun sprechen Tschadische Sprachen, die als ein alter Zweig der Afro-asiatischen Sprachfamilie eingestuft werden. Die ausgestorbene Sprache des Now der alten Ägypter gehörte auch der gleichen Sprachfamilie an. Personen, deren Y-Chromosomen alle Mutationen an den internen Nullpunkten des Y-DNA Baums aufweisen unter Einbeziehung von M207 (definiert Haplogruppe R), aber weder die Mutation M173 (definiert Haplogruppe R1) noch die Mutation M124 (definiert Haplogruppe R2) besitzen, werden Haplogruppe R* zugerechnet. Einige Fälle von Haplogruppe R* sind in den Proben der australischen Aborigines gefunden worden. Haplogruppe R* wurde auch in 10,3 % (10/97) einer Probe von Burusho und in 6,8 % (3/44) einer Probe von Kalash in Nordpakistan entdeckt. [2]
Untergruppen
Die Untergruppen der Haplogruppe R mit ihrer unterscheidenden Mutation, nach ihrer Gliederung, veröffentlicht 2010 von der International Society of Genetic Genealogy (ISOGG)[3]:
- R*
- R1 (M173/P241, M306/S1,P225, P231, P233, P234, P236, P238, P242, P245, P286, P294)
- R1a (L62/M513, L63/M511, L145/M449, L146/M420) Typisch für Bevölkerungen von Mittel- und Osteuropa, Zentralasien und Südasien, mit einer mittleren Verbreitung in Westeuropa, Südwestasien und Südsibirien
- R1a1 (L120/M516, L122/M448, M459, SRY1532.2/SRY10831.2)
- R1a1a (M17, M198, M417, M512, M514, M515)
- R1a1a1 (M56)
- R1a1a2 (M157)
- R1a1a3 (M64.2, M87, M204)
- R1a1a4 (P98)
- R1a1a5 (PK5)
- R1a1a6 (M434)
- R1a1a7 (M458)
- R1a1a7a (M334)
- R1a1a7b (L260)
- R1a1a7*
- R1a1a*
- R1a1*
- R1a1a (M17, M198, M417, M512, M514, M515)
- R1a*
- R1a1 (L120/M516, L122/M448, M459, SRY1532.2/SRY10831.2)
- R1b (M343) Typisch für Bevölkerungen in Westeuropa, mit einer mittleren Verbreitung außerhalb Eurasiens und in Teilen von Afrika
- R1b1 (P25, L278)
- R1b1a (V88)
- R1b1a1 (M18)
- R1b1a2 (V8)
- R1b1a3 (V35)
- R1b1a3a (V7)
- R1b1a3*
- R1b1a4 (V69)
- R1b1a*
- R1b1b (P297)
- R1b1b1 (M73)
- R1b1b2 (L265, M269, S3, S10, S13, S17)
- R1b1b2a (L23/S141, L49, L150)
- R1b1b2a1 (L51/S167)
- R1b1b2a1a (L11/S127, L52, L151, P310/S129, P311/S128)
- R1b1b2a1a1 (M405/S21/U106)
- R1b1b2a1a1a (M467/S29/U198)
- R1b1b2a1a1b (P107)
- R1b1b2a1a1c (DYS439(Null)/L1/S26)
- R1b1b2a1a1d (L48/S162)
- R1b1b2a1a1d1 (L47)
- R1b1b2a1a1d1a (L44)
- R1b1b2a1a1d1a1 (L45, L46, L164)
- R1b1b2a1a1d1a*
- R1b1b2a1a1d1*
- R1b1b2a1a1d1a (L44)
- R1b1b2a1a1d2 (L148)
- R1b1b2a1a1d*
- R1b1b2a1a1d1 (L47)
- R1b1b2a1a1e (L257)
- R1b1b2a1a1*
- R1b1b2a1a2 (P312/S116)
- R1b1b2a1a2a (M65)
- R1b1b2a1a2b (M153)
- R1b1b2a1a2c (M167/SRY2627)
- R1b1b2a1a2d (S28/U152)
- R1b1b2a1a2d1 (M126)
- R1b1b2a1a2d2 (M160)
- R1b1b2a1a2d3 (L2/S139)
- R1b1b2a1a2d3a (L20/S144)
- R1b1b2a1a2d3*
- R1b1b2a1a2d4 (L4)
- R1b1b2a1a2d*
- R1b1b2a1a2e (L165/S68)
- R1b1b2a1a2f (L21/S145)
- R1b1b2a1a2f1 (M37)
- R1b1b2a1a2f2 (M222/USP9Y+3636)
- R1b1b2a1a2f3 (P66)
- R1b1b2a1a2f4 (L226/S168)
- R1b1b2a1a2f5 (L193/S176)
- R1b1b2a1a2f*
- R1b1b2a1a2*
- R1b1b2a1a*
- R1b1b2a1a1 (M405/S21/U106)
- R1b1b2a1*
- R1b1b2a1a (L11/S127, L52, L151, P310/S129, P311/S128)
- R1b1b2a*
- R1b1b2a1 (L51/S167)
- R1b1b2*
- R1b1b2a (L23/S141, L49, L150)
- R1b1b*
- R1b1c (M335)
- R1b1*
- R1b1a (V88)
- R1b*
- R1b1 (P25, L278)
- R1*
- R1a (L62/M513, L63/M511, L145/M449, L146/M420) Typisch für Bevölkerungen von Mittel- und Osteuropa, Zentralasien und Südasien, mit einer mittleren Verbreitung in Westeuropa, Südwestasien und Südsibirien
- R2 (M124) Typisch für Bevölkerungen von Südasien, mit einer mittleren Verbreitung in Zentralasien und dem Kaukasus
R1
Haplogruppe R1 bestimmt die Mehrheit der Haplogruppe R in Form seiner Untergruppen, R1a and R1b.[4][5][6]
R1a
Die höchsten Konzentrationen von R1a (> 50 %) werden entlang der eurasischen Steppe gefunden: bei den Ishkashimi (68 %), den Tadschiken, der Bevölkerung von Khojant (64 %), Kirgisen (63,5 %), von Sorben (63,39 %), von Polen (56,4 %) und von Ukrainern (54,0 %) und im extremen Norden von Indien unter den Kaschmir Pandits (72 %). [7][8][9][5] [10]
R1a wurde von einzelnen Forschern in folgenden geschichtlichen Zusammenhängen gesehen:
- der Rückbesiedlung von Eurasien aus dem Refugium in der Ukraine Ende der letzten Eiszeit. [11][5]
- der Expansion der Kurgankultur von der Ponto-Kaspischen Steppe, die mit der Verbreitung der indoeuropäischen Sprachen einherging [8][5]
R1b
Ursprung
Die Y-Haplogruppe R1b ist eine Abzweigung von R1 (M173), und wird durch den M343 Marker unterschieden. [12] Der R1b Stamm scheint einen viel höheren Grad an innerer Verschiedenartigkeit/Diversität als R1a zu haben, was nahelegt, dass die Mutation M343, die R1b von R1* trennt, beträchtlich früher aufgetreten sein muss als die Mutation SRY1532, die R1a unterscheidet. Das Genographic Project (geleitet von der National Geographic Society), [4] nimmt an, dass R1b auf der Iberischen Halbinsel als Refugium der letzten Eiszeit entstanden ist, von wo aus sich die Gene danach wieder ausbreiteten.
Heutige Verbreitung
In Europa ist R1b (mit den Untergruppen R1b1 und R1b3, früher Hg1 und Eu18 benannt ) die häufigste Y-Haplogruppe. Die Konzentration erreicht in Teilen des nordwestlichen Irland 98 %; in Nord- und Westengland, Spanien[5] , Portugal und Irland [6] bis 90 % und im südöstlichen England und den Niederlanden [5] noch etwa 70 %. Darüber hinaus ist R1b in einigen Teilen Algeriens mit ca. 10 % vertreten.[13]
R2
90 % der R2 Träger werden auf dem Indischen Subkontinent gefunden. Sie wurde ebenfalls im Kaukasus und in Zentralasien entdeckt.
R2( M124) entstand vor ungefähr 25.000 Jahren im südlichen Zentralasien. Seine Träger wanderten nach Süden als Teil der zweiten[14] [15] großen Auswanderungswelle nach Indien. Die fast ausschließliche Existenz von R2 in Indien könnte auch bedeuten, dass die Verzweigung von R in R1 und in R2 in Mittelindien oder in Westindien geschah.
Literatur
- Luigi Luca Cavalli-Sforza: The History and Geography of Human Genes. Princeton University Press, 1994. ISBN 0-691-08750-4
- Semino et al.: The Genetic Legacy of Paleolithic Homo sapiens sapiens in Extant Europeans. 2000..
- Wells et al.: The Eurasian Heartland: A continental perspective on Y-chromosome diversity. 2001..
- Passarino et al.: Different genetic components in the Norwegian population revealed by the analysis of mtDNA and Y chromosome polymorphisms. 2002..
- Behar et al.: Multiple Origins of Ashkenazi Levites: Y Chromosome Evidence for Both Near Eastern and European Ancestries. 2003..
- Saha Anjana, Sharma Swarkar, Bhat Audesh,Pandit Awadesh, Bamezai Ramesh (2005). Genetic affinity among five different population groups in India reflecting a Y-chromosome gene flow. J Hum Genet;50:49–51 PMID 15611834
- Sanghamitra Sengupta et al. (2006), Polarity and Temporality of High-Resolution Y-Chromosome Distributions in India Identify Both Indigenous and Exogenous Expansions and Reveal Minor Genetic Influence of Central Asian Pastoralists, American Journal of Human Genetics, 78:202-221
- C. Cinnioglu et al. (2004), Excavating Y-chromosome haplotype strata in Anatolia, Human Genetics 114(2):127-48.
Siehe auch
Weblinks
- ISOGG tree of haplogroup R
- R branch of the haplotree
- 2005 Y-Chromosome Phylogenetic Tree
- World and European haplogroup prevalence maps (PDF-Datei; 386 kB)
- Spread of Haplogroup R1, from The Genographic Project, National Geographic (18. September 2010 disabled / offline)
- Spread of R1a1, from the Genographic Project, National Geographic (18. September 2010 disabled / offline)
- Spread of R1b, from the Genographic Project, National Geographic
- World Families Network zu R1b
- World Families Network zu R1a
- Worldfamilies.net Page on Haplogroups
Einzelnachweise
- ↑ Mulcare, Charlotte A. et al: The T Allele of a Single-Nucleotide Polymorphism 13.9 kb Upstream of the Lactase Gene (LCT) (C513.9kbT) Does Not Predict or Cause the Lactase-Persistence Phenotype in Africans. In: The American Journal of Human Genetics. 74. Jahrgang, 2004, S. 1107 (uchicago.edu [PDF]).
- ↑ Sadaf Firasat, Shagufta Khaliq, Aisha Mohyuddin, Myrto Papaioannou, Chris Tyler-Smith, Peter A Underhill and Qasim Ayub, "Y-chromosomal evidence for a limited Greek contribution to the Pathan population of Pakistan," European Journal of Human Genetics (2007) 15, 121–126.
- ↑ http://www.isogg.org/tree/ ISOGG Website
- ↑ 4,0 4,1 Haplogroup R1 (M173). In: The Genographic Project. National Geographic Society. Abgerufen am 11. März 2008.
- ↑ 5,0 5,1 5,2 5,3 5,4 5,5 Semino et al. 2000
- ↑ 6,0 6,1 |Rosser et al.2000
- ↑ High-Resolution Phylogenetic Analysis of Southeastern Europe Traces Major Episodes of Paternal Gene Flow Among Slavic Populations - Pericic et al. 22 (10): 1964 - Molecular Bi...
- ↑ 8,0 8,1 Wells et al. 2001
- ↑ Behar et al. 2003
- ↑ The Autochthonous Origin and a Tribal Link of Indian Brahmins: Evaluation Through Molecular Genetic Markers, by S. Sharma (1,2), E. Rai (1,2), S. Singh (1,2), P.R. Sharma (1,3), A.K. Bhat (1), K. Darvishi (1), A.J.S. Bhanwer (2), P.K. Tiwari (3), R.N.K. Bamezai (1) 1) NCAHG, SLS, JNU, New delhi; 2) Department of Human Genetics, GNDU, Amritsar; 3) Centre for Genomics, SOS zoology, JU, Gwalior, Page 273 (1344/T), Published in THE AMERICAN SOCIETY OF HUMAN GENETICS 57th Annual Meeting, October 23–27, 2007, San Diego, California
- ↑ Passarino et al. 2002
- ↑ Note that in earlier literature the M269 marker, rather than M343, was used to define the R1b haplogroup. Then, for a time (from 2003 to 2005) what is now R1b1c was designated R1b3.
- ↑ Analysis of Y-chromosomal SNP haplogroups and STR haplotypes in an Algerian population sample
- ↑ The first consisted of African migrants who traveled along the Indian coastline some 50,000 to 60,000 years ago.
- ↑ The Genographic Project. National Geographic Society. Abgerufen am 13. März 2008.
| Evolutionsbaum Haplogruppen Y-chromosomale DNA (Y-DNA) 2012 | |||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Adam der Y-DNA | |||||||||||||||||||||||||||||||
| A0 | A1 | ||||||||||||||||||||||||||||||
| BT | |||||||||||||||||||||||||||||||
| B | CT | ||||||||||||||||||||||||||||||
| CF | DE | ||||||||||||||||||||||||||||||
| C | F | D | E | ||||||||||||||||||||||||||||
| G | H | IJK | |||||||||||||||||||||||||||||
| IJ | K | ||||||||||||||||||||||||||||||
| I | J | LT | MNOPS | ||||||||||||||||||||||||||||
| L | T | M | NO | P | S | ||||||||||||||||||||||||||
| N | O | Q | R | ||||||||||||||||||||||||||||