Segmentierungsgen
Segmentierungsgene bestimmen während der Embryogenese von Insekten die Anzahl und innere Organisation der Segmente. Erforscht wurden sie am Modellorganismus Drosophila melanogaster, vor allem durch Analyse der Gene mutierter Fliegen oder deren Embryonen, die Missbildungen der Segmentation oder der Körpergliederung zeigten. Die Gene tragen Namen, die in der Regel von Mutationen abgeleitet sind, die zu ihrer Entdeckung führten. Genprodukt der Segmentierungsgene sind Proteine mit regulatorischen Aufgaben, die sich an die DNA anlagern und so andere Gene quasi wie ein Schalter ein- und ausschalten, diese werden Transkriptionsfaktoren genannt. Dazu besitzen die Zielgene Sequenzen, die am DNA-Strang vor dem proteincodierenden Genabschnitt angeordnet sind und selbst nicht transkribiert werden. Da das vordere Ende eines DNA-Strangs als "cis"-Ende bezeichnet wird (das hintere als "trans"), spricht man von "cis-regulatorischen" Abschnitten oder Cis-Elementen. Die Transkriptionsfaktoren der Segmentierungsgene sind in einer regulatorischen Kaskade hintereinandergeschaltet, das bedeutet, dass übergeordnete, früh ausgeprägte Segmentierungsgene die späteren je nach ihrer Lage im sich entwickelnden Embryo ein- oder ausschalten. Es entwickelt sich so ein Streifenmuster aus hintereinander angeordneten Streifen, in denen jeweils ein bestimmtes Segmentierungsgen (oder eine Gruppe von solchen) aktiv ist. Durch diese Zellstreifen wird die spätere Segmentierung des Körpers vorgebildet. Die Zellen, die das Körpergewebe selbst aufbauen, erhalten durch die Segmentierungsgene so eine Information über ihre Lage im sich entwickelnden Organismus. Je nach Lage können sie wachsen, sich teilen und differenzieren oder auch absterben (Programmierter Zelltod oder Apoptose).
Spätere Analysen haben ergeben, dass homologe Gene zahlreicher der Segmentierungsgene im gesamten Tierreich bei allen daraufhin untersuchten Organismen vorkommen, sie organisieren überall die Bildung der Körperachsen. Dies erfolgt bei nicht segmentierten Organismen in sehr ähnlicher und analoger Weise. Andere der Segmentierungsgene sind nur bei Insekten oder Arthropoden ausgeprägt, einige sogar nur bei den Zweiflüglern (Dipteren), einzelne sind sogar ausschließlich von Drosophila bekannt.
Die Regulationskaskade
Der Embryo der Taufliege Drosophila entwickelt sich aus der Eizelle, indem eine äußere Zellschicht, das Blastoderm, einen zentralen Dottervorrat umwächst. Innerhalb des Blastoderms differenziert sich ein Keimstreifen. Bei den ersten Teilungsschritten teilen sich nur die Zellkerne, während das Zytoplasma nicht durch Zellmembranen geteilt wird, ein solcher ungeteilter Verband wird Syncytium genannt. Morphologisch ist in diesem Stadium keine Gliederung erkennbar, das Vorder- und das Hinterende des entstehenden Embryos sehen gleich aus. Durch Experimente in den 1960er und 1970er Jahren konnte gezeigt werden, dass die Körperlängsachse in diesem Stadium allerdings schon festgelegt ist. Es zeigte sich, dass der organisierende Faktor, der die Körperlängsachse festlegt, dem Ei bereits von der Mutter mitgegeben wird. Dies beruht auf Substanzen, die in den vorderen bzw. hinteren Eipolen konzentriert sind. Mutationen dieser Gene führen zu schweren Missbildungen, z. B. wird statt der Anlage der vorderen Körperhälfte die hintere Hälfte doppelt angelegt. Da die entsprechenden Genprodukte (Transkripte) von der Mutter stammen, werden die dazugehörigen Gene maternale Gene genannt. Die ins Cytoplasma des Eis übertragenen Produkte der maternalen Gene legen also das Vorder- und Hinterende des Embryos fest. Ihre Proteine bilden im Embryo ein Konzentrationsgefälle (einen Gradienten) aus, bei dem einige am Vorderende in der höchsten Konzentration auftreten, einige am Hinterende. Die dazwischen liegenden Zellen enthalten von beiden jeweils unterschiedliche Anteile, je nachdem, wie weit vorn bzw. hinten sie liegen. Je nach Konzentration der Proteine der maternalen Gene wird im Embryo dann eine Gruppe weiterer Gene aktiviert, die Lückengene (häufiger, aus dem Englischen übernommen, Gap-Gene) genannt werden. Die Namensgebung beruht darauf, dass bei der Mutation in einem dieser Gene dem Embryo ganze Körperabschnitte fehlen. Bei Drosophila existieren fünf Gap-Gene. Jedes Gap-Gen aktiviert nun je nach Lage ein anderes Gen einer weiteren Klasse, der Paarregel-Gene (engl. pair rule gene). Der Name beruht hier darauf, dass bei einer Mutation der Embryo jeweils nur die halbe Segmentzahl aufweist. Dadurch wird der Embryo in sieben Streifen organisiert. Die Paarregel-Gene aktivieren als nächste Klasse die Segmentpolarisations-Gene. Jeder Streifen wird dabei in zwei Teilstreifen geteilt. Dadurch sind die vierzehn Körpersegmente der Fliegenmade vorgebildet. (Tatsächlich ist die Sache etwas verwickelter: Es werden sog. Parasegmente vorgebildet, die jeweils aus dem Vorderende eines und dem Hinterende des benachbarten Segments bestehen).
Im fertigen Organismus sind diese Segmente allerdings untereinander nicht morphologisch gleich, sondern differenzieren sich in Körperabschnitte (Tagmata): Kopf, Rumpf (Thorax) und Hinterleib (Abdomen). Je nach Lage werden unterschiedliche Anhänge und andere Organe wie Antennen, Beine etc. ausgebildet oder nicht. Diese Identität wird den Segmenten durch eine weitere Genklasse zugewiesen, die Hox-Gene. Bei der Taufliege gibt es acht Hox-Gene. Die Expression der Hox-Gene ist dabei nicht genau an die Segmentabfolge geknüpft. Einige Körpersegmente exprimieren dasselbe Hox-Gen, in anderen werden zwei davon exprimiert.
Insgesamt sind ca. 40 bis 50 Gene identifiziert, die an dieser Musterbildung beteiligt sind, die Rolle einiger davon ist noch unklar. Eine ähnliche Signalabfolge, an der ebenfalls maternale und embryonale Gene beteiligt sind, legt die Dorsal-ventral-Achse des Embryos fest, d. h. bestimmt über oben (Rückenseite) und unten (Bauchseite). Diese Gene sind später entdeckt worden und insgesamt weniger gut bekannt.
Maternale Gene
Die maternalen Gene sind, wie der Name andeutet, im mütterlichen Organismus aktiv. Ihre Genprodukte, meist RNA (Transkripte), seltener bereits fertige Proteine, werden dem sich entwickelnden Ei mitgegeben. Die Gene caudal (cad) und hunchback (hb) besitzen eine Doppelrolle, sie werden sowohl maternal wie auch später im Embryo selbst transkribiert. Wichtigstes der maternalen Gene, die die Orientierung der Körperlängsachse vorgeben, ist bicoid (bcd). Das Bicoid-Protein legt das Vorderende des Embryos fest und aktiviert je nach Konzentration andere Gene in nach hinten fortschreitenden Zonen unterschiedlicher Breite. cad und hb sind zunächst im Ei nahezu gleich verteilt. Da aber Bcd ihre Expression hemmt, reichern sie sich im hinteren Bereich an. Im hinteren Eipol ist eine weitere Gruppe von RNAs und Proteinen angereichert.
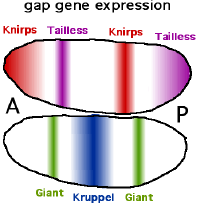
Lückengene
Der Name Lückengen (engl. Gap-Gen) rührt daher, dass ein Funktionsausfall dieser Gene zu Lücken in der Segmentierung, dem Fehlen von Körpersegmenten, führt.[1][2] Sie sind für die Aufteilung in einen vorderen, mittleren und hinteren Bereich zuständig.[2] Zu den derzeit elf bekannten Lückengene zählen giant (gt oder gat), hunchback (hb), knirps (kni), Krüppel (im engl. meist "kruppel") (kr) und tailless (tll). cad und hb haben eine Doppelrolle. Sie werden sowohl maternal (von mütterlicher Herkunft) als auch im Embryo selbst exprimiert. Die Gap-Gene werden initial relativ weit verteilt exprimiert und durch Selbstorganisation später auf die passenden Streifen beschränkt. Wie auch die Produkte der maternalen Gene sind die exprimierten Transkriptionsfaktoren nur eine kurze Zeit aktiv. Ihre Aktivität endet, sobald sich das Muster herausgebildet hat und die folgenden Schritte initiiert sind, das ist schon nach etwa zwei Stunden Entwicklungszeit der Fall. Im späteren Organismus spielen sie keine Rolle mehr, teilweise sind sie allerdings an weiteren Musterbildungsprozessen unabhängig von ihrer Rolle bei der Festlegung der Körperachse beteiligt.
Die Expression der Gap-Gene wird von einer Kombination von maternalen Genen und Wechselwirkungen der Gap-Gene untereinander reguliert. Außerdem spielt das an den Zellenden, nicht aber dazwischen exprimierte Gen torso (tor) eine Rolle. Torso codiert einen Transmembran-Rezeptor, der von Substanzen in der Eihülle aktiviert wird. Das maternale Bcd-Protein schaltet das hb-Gen in der vorderen Körperhälfte ein, so dass zwei scharf geschiedene Hälften entstehen (Alles-oder-Nichts Reaktion). Gleichzeitig unterdrückt Bcd die Transkription von cad, so dass das Cad-Protein nur in der hinteren Körperhälfte vorkommt. Die anderen Gap-Gene bilden in ähnlicher Weise einen oder zwei Streifen an verschiedenen Stellen des Embryos aus. So wird Kr vor allem in einer Region nahe der Mittellinie gebildet, indem es von Bcd aktiviert, aber von Hb reprimiert (verhindert) wird. Kni wird durch einen vergleichbaren Mechanismus am Vorderende und in einem Streifen im hinteren Abschnitt exprimiert.
Die verschiedenen Körperabschnitte sind so in der Regel durch eines oder durch eine Kombination von jeweils zwei Gap-Proteinen charakterisiert. Die gap-Proteine zeigen in der Regel zu einem Maximum ansteigende und dahinter abfallende Konzentrationen, deren Bereiche sich mehr oder weniger breit überlappen.
Paarregel-Gene
Die embryonalen Streifen, die durch die Gap-Gene vorgegeben sind, werden jeweils durch verschiedene Kombinationen der Paarregel-Gene spezifiziert. Die Paarregel-Gene even-skipped (eve) und fushi tarazu (ftz) werden jeweils abwechselnd in je sieben Streifen exprimiert. Andere Gene wie runt (run) und hairy (h) zeigen ähnliche Muster. Das aperiodische Streifenmuster der Gap-Gene wird so durch ein periodisches Muster überlagert. Die Konzentrationen der Paarregel-Proteine sind im Endzustand je nach Zellage scharf geschieden, sie überlappen nicht mehr wie diejenigen der Gap-Gene. Mutationen bei Paarregel-Genen bewirken den Verlust jedes zweiten Segmentes, Paarregel-Gene kontrollieren also die Ausbildung der geradzahligen oder ungeradzahligen Segmente.[3]
Segmentpolaritätsgene
Die Segmentpolaritätsgene legen sowohl die endgültige Abfolge der (Para-)Segmente wie auch ihre Polarität, d. h. ihr Vorder- und Hinterende, fest. Das Segmentpolaritätsgen Engrailed (en) wird in einer schmalen Zone nahe dem Vorderende von vierzehn Parasegmentstreifen exprimiert. Hedgehog (hh) zeigt ein ähnliches Muster. Das Gen Wingless (wn) wird dagegen in Streifen nahe dem Hinterende der Parasegmentstreifen aktiv. Dieses Muster gliedert den Keimstreifen des Embryos in gleichartige Streifen, die allerdings nicht synchron, sondern nacheinander erzeugt werden. Im Gegensatz zu den vorangehenden Segmentierungsgenen bleiben die Segmentpolaritätsgene lange Zeit, im Falle von Engrailed bis zum geflügelten Insekt (Imago), aktiv. Mit der Aktivität der Segementpolaritätsgene ist die Segmentierung abgeschlossen. In den folgenden Entwicklungsstadien wird die Identität der verschiedenen Segmente weiter spezifiziert. Dies ist vor allem die Aufgabe der Hox-Gene. Bei der Aktivierung der spezifischen Hox-Gene spielen die Gap-Gene (die, im Gegensatz zu den späteren Stadien der Segmentierungskaskade, nicht periodisch sind) direkt eine wichtige Rolle.
Auftreten bei anderen segmentierten Tieren
Das am Modellorganismus Drosophila entdeckte grundsätzliche Schema konnte bei späteren Forschungen in Grundzügen bei allen daraufhin untersuchten Arthropoden gefunden werden. Die Segmentbildung läuft bei allen Arten über eine Regulationskaskade, über ein von den Segmentpolaritätsgenen in Segmente gegliedertes Keimband, ab. Die davor liegenden Entwicklungsstadien sind allerdings bei anderen Arthropoden im Detail verschieden. Dies liegt z. B. daran, dass die meisten Arthropoden, anders als Drosophila, ihre Entwicklung nicht mit einem Syncytium beginnen. Außerdem werden bei sehr vielen Arten die Segmente nicht gleichzeitig angelegt, sondern im Laufe der Ontogenese an einer Segmentbildungszone am Hinterende erst nach und nach gebildet. Bei den relativ wenigen bisher näher untersuchten Arten konnte bicoid (oder ein homologes Gen) bisher nur bei anderen Zweiflüglern (Dipteren) gefunden werden. Bei anderen Arten sind zwar auch maternale Gene an der Gliederung beteiligt, aber im Detail jeweils andere. Die Gene cad und nanos (nos) scheinen dabei weit verbreitet zu sein. Orthologe oder homologe Gene zu den meisten Segmentbildungsgenen von Drosophila sind in den meisten Arthropoden gefunden worden, allerdings scheint ihre Rolle im Detail jeweils verschieden zu sein. Bei den Spinnentieren und den Tausendfüßern wurden darüber hinaus zusätzlich andere Regulationsmuster gefunden, die der Musterbildung der Somite bei Wirbeltieren ähnlicher sind.
Literatur
- Michael Akam (1987): The molecular basis for metameric pattern in the Drosophila embryo. Development 101: 1-22.
- Dmitri Papatsenko, Michael Levine (2011): The Drosophila Gap Gene Network Is Composed of Two Parallel Toggle Switches. PLoS ONE 6(7): e21145. doi:10.1371/journal.pone.0021145
- Andrew D. Peel, Ariel D. Chipman, Michael Akam (2005): Arthropod segmentation: beyond the Drosophila paradigm. Nature Review Genetics doi:10.1038/nrg1724
Einzelnachweise
- ↑ C.R. Bartram et al.: Humangenetische Diagnostik: Wissenschaftliche Grundlagen und gesellschaftliche Konsequenzen. Springer, 2000, ISBN 978-3540679455, S. 30.
- ↑ 2,0 2,1 Jan Zravý, David Storch, Stanislav Mihulka: Evolution: Ein Lese-Lehrbuch. Spektrum Akademischer Verlag, 2009, ISBN 978-3827419750, S. 230.
- ↑ Monica Hirsch-Kauffmann, Manfred Schweiger: Biologie und molekulkare Medizin für Mediziner und Naturwissenschaftler. Thieme Verlag, 2009, ISBN 978-3137065074, S. 254.