SARS-CoV-2
- Seiten mit Skriptfehlern
- Wikipedia:Vorlagenfehler/Vorlage:Literatur/Interner Fehler
- Wikipedia:Vorlagenfehler/Vorlage:Literatur/Abrufdatum
- Wikipedia:Veraltet nach Februar 2021
- Virussubtyp
- Meldepflichtiger Erreger
- COVID-19-Pandemie
| SARS-CoV-2 | ||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ||||||||||||||||||||||||||
| Systematik | ||||||||||||||||||||||||||
| ||||||||||||||||||||||||||
| Taxonomische Merkmale | ||||||||||||||||||||||||||
| ||||||||||||||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||||||||||||||
| {{Modul:Vorlage:lang}} Modul:Multilingual:149: attempt to index field 'data' (a nil value) | ||||||||||||||||||||||||||
| Kurzbezeichnung | ||||||||||||||||||||||||||
| SARS-CoV-2[1] | ||||||||||||||||||||||||||
| Links | ||||||||||||||||||||||||||
|
SARS-CoV-2 (Abk. für englisch severe acute respiratory syndrome coronavirus 2,[1] deutsch Schweres-akutes-Atemwegssyndrom-Coronavirus 2[6], umgangssprachlich nur (neues) Coronavirus genannt; vormals auch „2019-nCoV“, „{{Modul:Vorlage:lang}} Modul:Multilingual:149: attempt to index field 'data' (a nil value)“, „neuartiges Coronavirus 2019“ sowie „Wuhan-Coronavirus“[7]) ist ein im Dezember 2019 in der chinesischen Stadt Wuhan, Provinz Hubei, neu identifiziertes Virus der Virusfamilie Coronaviridae.[8] Das Virus verursacht die neue Atemwegserkrankung COVID-19[9][10] und war Auslöser der COVID-19-Pandemie, die von der Weltgesundheitsorganisation (WHO) am 30. Januar 2020 als „gesundheitliche Notlage von internationaler Tragweite“ und am 11. März 2020 als Pandemie eingestuft wurde. Das Virus wird hauptsächlich durch Tröpfcheninfektion übertragen.
Entdeckungsgeschichte
Im Dezember 2019 wurde in der Stadt Wuhan eine Häufung schwerer Lungenentzündungen mit unbekannter Ursache festgestellt.[11] Am 30. Dezember 2019 informierte der chinesische Arzt Li Wenliang in einer WeChat-Gruppe seine Arztkollegen über sieben Patienten, die mit Verdacht auf eine Infektion mit dem SARS-Virus im Zentralkrankenhaus Wuhan behandelt wurden;[12] dafür wurde er von der chinesischen Polizei ermahnt. Li Wenliang erkrankte später selber an COVID-19 und verstarb an den Folgen.[13] Das Chinesische Zentrum für Krankheitskontrolle und -prävention entsandte am 31. Dezember 2019 ein Team in die Stadt.[14] Am selben Tag wurde das China-Büro der Weltgesundheitsorganisation (WHO) durch die chinesischen Behörden offiziell darüber informiert, dass im Dezember 2019 in Wuhan mehrere Personen an einer schweren Lungenentzündung erkrankt waren und dass als deren Ursache ein bis dahin uncharakterisierter, infektiöser Erreger vermutet werde. Bis zum 3. Januar 2020 wurden der WHO insgesamt 44 Erkrankte gemeldet, darunter mehrere Schwerkranke. Da mehrere Erkrankte auf dem örtlichen wet market „Wuhan Huanan Großhandelsmarkt für Fische und Meeresfrüchte“ (chinesisch {{Modul:Vorlage:lang}} Modul:Multilingual:149: attempt to index field 'data' (a nil value), Pinyin {{Modul:Vorlage:lang}} Modul:Multilingual:149: attempt to index field 'data' (a nil value)) als Verkäufer oder Händler arbeiteten, wurde in diesem Markt der primäre Infektionsort vermutet.[15][16] Kurz nach dem Auftreten der Krankheit im Dezember 2019 hatten 27 und damit 66 % der ersten 41 Krankenhauspatienten in Wuhan den Markt im Zentrum der Stadt besucht. Allerdings standen die Infektionen der übrigen betroffenen Personen laut dieser Studie in keinerlei Zusammenhang zu diesem Ort.[17]
Am 7. Januar 2020 gab der mit der Virusidentifizierung befasste leitende chinesische Virologe Xu Jianguo ({{Modul:Vorlage:lang}} Modul:Multilingual:149: attempt to index field 'data' (a nil value)) bekannt, dass es sich bei dem Krankheitserreger um ein bis dahin unbekanntes Coronavirus handle. Dies hätten Untersuchungen von Blutproben und Rachenabstrichen von 15 Erkrankten ergeben. In einer Stellungnahme der WHO am 9. Januar 2020 wurde diese Erkenntnis bestätigt.[18][19] Am 13. Januar 2020 wurde die komplette Genomsequenz eines Isolats des neuen Coronavirus in der NCBI-GenBank hinterlegt (GenBank-Nummer MN908947).[20] Nahezu gleichzeitig wurde ein erstes Nachweisverfahren publiziert.[21][22][23]
Eine inzwischen vorgenommene phylogenetische Analyse der Genomsequenzen von Umweltproben (etwa von Oberflächen) des Marktes zeigte, dass sie mit den Viren der ersten Patienten aus Wuhan sehr nahe verwandt sind.[24] Nach einer Studie des Wuhan Hospitals hatte der erste identifizierte Patient den Markt allerdings nicht besucht.[25] Es zeigte sich also, dass nicht alle frühen COVID-19-Fälle mit dem Markt in Verbindung gebracht werden können und die Historie des Ausbruchs wohl komplizierter ist als zunächst angenommen.[24]
Benennung
In der Öffentlichkeit wird das Virus „SARS-CoV-2“ meist (nach der Virusfamilie) als „neuartiges Coronavirus“,[26] „neues Coronavirus“,[27] „Coronavirus“, nur „Corona“ oder gelegentlich (nach der Krankheit) als „Covid-19-Virus“ bezeichnet.[28][6]
Die von der Weltgesundheitsorganisation (WHO) vom 13. Januar bis 11. Februar 2020 verwendete Bezeichnung „2019-nCoV“ galt nach Aussage der WHO nur als „vorläufig“.[29] Das erste sequenzierte Virusisolat wurde in der Erstbeschreibung als WH-Human-1 coronavirus (WHCV) bezeichnet (WH = Wuhan) und als Wuhan seafood market pneumonia virus isolate Wuhan-Hu-1 in die NCBI-Taxonomie-Datenbank (die für Virusnamen und -klassifikationen nicht maßgebend ist) aufgenommen. Das Virus wurde dort danach – ebenfalls vorläufig – als Wuhan seafood market pneumonia virus geführt; als Synonyme wurden 2019-nCoV und Wuhan coronavirus erwähnt.[30] Die WHO griff diverse Namensvorschläge nicht auf, die allesamt gemeinsam hatten, das Virus – in Anlehnung an MERS-CoV (Middle East respiratory syndrome coronavirus) – nach dem Ort seiner Erstidentifikation (der Stadt Wuhan) als Wuhan respiratory syndrome coronavirus (WRS-CoV) zu benennen. Ein von Fachleuten genannter Grund waren Beschwerden in der Vergangenheit, als Viren ihren Namen nach einzelnen Ländern oder Regionen erhalten hatten[31] (beispielsweise das Marburg-Virus). Die WHO hatte daher 2015 Empfehlungen herausgegeben, wie neue Krankheitserreger und Erkrankungen zu benennen seien. Eine Benennung nach dem Entdeckungsort wurde dabei explizit für unerwünscht erklärt.[32] In der NCBI-Taxonomie-Datenbank aufgeführte Synonyme sind 2019-nCoV, COVID-19, COVID-19 virus, Wuhan coronavirus und Wuhan seafood market pneumonia virus (Stand 16. Februar 2020).[33]
Am 11. Februar 2020 gab die WHO bekannt, die durch das Virus verursachte Erkrankung als „COVID-19“ bzw. „Covid-19“ (für corona virus disease 2019) benannt zu haben.[10][34] Am selben Tag wurde von der Coronavirus Study Group (CSG) des International Committee on Taxonomy of Viruses (ICTV) auf dem Preprint-Server bioRxiv für das Virus die Bezeichnung SARS-CoV-2 (für severe acute respiratory syndrome coronavirus 2) vorgeschlagen.[1] Dem widersprach eine Woche später eine Gruppe chinesischer Virologen, die stattdessen als Bezeichnung human coronavirus 2019, HCoV-19 („Humanes Coronavirus 2019“) vorschlug. Als Begründung nannten sie die Ähnlichkeit dieser Virusbezeichnung mit der Bezeichnung der von der WHO als COVID-19 benannten Krankheit und die Gefahr der Verwechslung von SARS-CoV-2 mit SARS-CoV in der Öffentlichkeit. Sie betonten, dass sich „2019-nCoV“ von dem SARS-Virus in biologischer und epidemiologischer Hinsicht unterscheide, ebenso wie die klinischen Symptome von COVID-19 und SARS verschieden seien.[35] Zur Unterscheidung wird der Erreger von SARS auch als SARS-CoV-1 bezeichnet.[36]
Merkmale
Systematik
SARS-CoV-2 ist einer der Vertreter der Spezies Severe acute respiratory syndrome-related coronavirus (SARS-assoziiertes Coronavirus, Akronym SARSr-CoV). Zu dieser Spezies gehören aktuell (März 2020) nur SARS-CoV-2 und SARS-CoV-1. Letzteres war bisher einfach als SARS-CoV bekannt und ist der Erreger von SARS, während SARS-CoV-2 COVID-19 auslöst.
Die Spezies Severe acute respiratory syndrome-related coronavirus ist aktuell die einzige Spezies in der Untergattung Sarbecovirus. Der Name „Sarbecovirus“ bezieht sich auf die englische Bezeichnung {{Modul:Vorlage:lang}} Modul:Multilingual:149: attempt to index field 'data' (a nil value) (bzw. {{Modul:Vorlage:lang}} Modul:Multilingual:149: attempt to index field 'data' (a nil value)).[37][38]
Die Untergattung Sarbecovirus gehört der heutigen Gattung Betacoronavirus an. Die frühere Gattung Coronavirus wurde abgeschafft und deren Mitglieder auf die neuen Gattungen Alpha-, Beta-, Gamma- und Deltacoronavirus aufgeteilt. Der Name „Coronavirus“ kann heute informell alle Viren der Familie Coronaviridae bezeichnen und wird besonders gegenüber der breiten Öffentlichkeit zum einfacheren Verständnis gebraucht.
Das im Zusammenhang mit SARS-CoV-2 oft genannte Virus MERS-CoV gehört ebenfalls zur Gattung Betacoronavirus, dort aber zur Untergattung Merbecovirus.
Die Gattung Betacoronavirus gehört zur Unterfamilie der Orthocoronavirinae (früher schlicht Coronavirinae), und diese zur Familie der Coronaviridae, diese zur Unterordnung Cornidovirineae, diese zur Ordnung Nidovirales. Letztere wird noch in den virologischen Bereich der RNA-Viren bzw. Riboviren (Riboviria) klassifiziert, da ihr Erbmaterial aus RNA besteht. Dadurch werden aber keine weiteren Verwandtschaftsbeziehungen im phylogenetischen Sinne ausgedrückt.
Einen Stammbaum der SARS-CoV-2-Isolate, der ihre Verwandtschaft untereinander zeigt, findet man bei Li et al. (Ende Februar 2020).[39] Die bis dato gefundenen Isolate gliedern sich in zwei Hauptgruppen (L-Typ nach der Aminosäure Leucin und S-Typ nach Serin), was Anlass zur Vermutung gab, das Virus könnte sich in zwei (unterschiedlich infektiöse) Zweige aufgeteilt haben.[40][41] Allerdings war es nach Meinung anderer Experten zum Zeitpunkt Anfang März 2020 noch zu früh, darüber eindeutige Aussagen machen zu können.[42][43][44][45] Die in beiden Hauptzweigen des Stammbaums basal liegenden Isolate stammen aus Wuhan (Provinz Hubei), was zeigt, dass nach aktueller Datenlage das Virus dort seinen Ausgang genommen hat. Das bedeutet natürlich nicht, dass es unbekannte Vorläufer von anderswo, etwa der chinesischen Yunnan, in Tieren oder Menschen gegeben haben könnte; auch ein Einschleppen nach China durch den Import von Wirtstieren ist nicht ausschließbar (siehe unten →Herkunft und Wirtsspektrum). Eine weitere Studie Anfang April machte zu diesem Zeitpunkt dann drei Stämme A, B und C aus. Stamm A ist dem Fledermausvirus BatCoV/RaTG13 am ähnlichsten und scheint sich von Wuhan aus weltweit verbreitet zu haben, in Festlandchina selbst ist aber der Stamm B vorherrschend, der außer in China auch in Ostasien verbreitet ist. Stamm C ist der hauptsächliche Typ in Europa.[46][47]
Das Virus mutiert offenbar relativ langsam (ein bis zwei Mutationen pro Monat), es lässt sich also im Vergleich mit Influenzaviren zwei- bis viermal soviel Zeit.[48] Das bedeutet einerseits, dass es per Genomanalyse keine sehr hohe Auflösung bezüglich der Ausbreitungswege des Virus gibt, andererseits lässt es darauf hoffen, dass eine nach überstandener Krankheit erworbene Immunität lange (monatelang) anhält. Allerdings hatten isländische Virologen von deCODE Genetics (Vorlage:IsS) bis zum 24. März 2020 vierzig verschiedene Mutationen allein bei Infizierten aus diesem Land identifiziert.[49][50][51] Eine der betroffenen Personen war mit zwei verschiedenen Ausprägungen von SARS-CoV-2 coinfiziert.[52][53]
Bei der Überwachung der genetischen Vielfalt und der Entwicklung des Virus müssen unterschieden werden:
- Stille Mutationen, die sich wegen der Degeneration des Genetischen Codes nicht auf die kodierten Proteine auswirken und eine molekulargenetische Uhr definieren,
- Mutationen mit Auswirkungen auf den Phänotyp (das Erscheinungsbild des Virus in all seinen Ausprägungen). Diese weisen offenbar bei SARS-CoV-2 auf fortlaufende Anpassung an seinen neuen menschlichen Wirt hin. Wichtig für die Entwicklung von Antikörpern und Impfstoffen ist es herauszufinden, welche Teile der kodierten Proteine stabil bleiben und konserviert werden, damit die Mittel durch Anpassung der Viren nicht schnell wirkungslos werden.[54]
Eine genaue Analyse dieser Sachverhalte findet sich bei Lucy van Dorp et al. (2020),[54] siehe auch Ärzteblatt vom 6. Mai 2020: die Infektiosität des Virus könnte sich mit der Zeit erhöhen,[55] eine Befürchtung, die grundsätzlich auch beim bisher nur schwer von Mensch zu Mensch übertragbaren MERS-CoV besteht. Eine Zusammenfassung auf deutsch findet sich auf scinexx.de vom 8. Mai 2020.[56]
Die im Westen dominierende Form des Virus, die sich ab Februar 2020 in Europa stark ausbreitete und von dort auch in andere Länder, hat eine Mutation D614G im Spike-Protein[57] und weicht damit von der Wuhan-Variante ab, was Auswirkungen auf die Impfstoffentwicklung hat, die generell die Wuhan-Variante zugrundelegt.
Molekulargenetik und Phylogenetik
Das Virusgenom besteht, wie in Coronaviren üblich, aus einzelsträngiger RNA (ssRNA) mit positiver Polarität. Das Isolat Wuhan-Hu-1 (NCBI-GenBank-Nummer MN908947[58]) umfasst 29.903 nt (Nukleotide) mit zwei 265 nt bzw. 229 nt langen untranslatierten Bereichen am 5′-Ende bzw. am 3′-Ende.[20] Die putativen (vermuteten) Gene könnten für zehn Proteine codieren: ein 7096 Aminosäuren (AS) langes ORF1ab-Polyprotein (Replikase-Komplex), ein 1273 AS langes Oberflächen-Glykoprotein (S für englisch spikes, vergleiche Peplomer), ein 75 AS langes Hüllprotein (E für engl. envelope, vergleiche Virushülle), ein 222 AS langes Membran-Glykoprotein (M), ein 419 AS langes Nukleokapsid-Phosphoprotein (N) und weitere fünf Proteine (ORF3a, ORF6, ORF7a, ORF8 und ORF10).[20] Die Abfolge der Gene entspricht jener des SARS-Virus und der aller Coronaviren.[59]
Mit Stand 16. Februar 2020 gab es mehr als 40 vollständige Genomanalysen von SARS-CoV-2-Isolaten. Die Genomgröße liegt zwischen 29.825 und 29.903 nt.[58] Der GC-Gehalt (der Anteil der Nukleinbasen Guanin und Cytosin) liegt bei 38,0 Mol-Prozent.[60][61] Die beiden Virusisolate HKU-SZ-002a (NCBI-GenBank-Nummer MN938384[58]) und HKU-SZ-005b (NCBI-GenBank-Nummer MN975262[58]) stammen von Patienten einer Familie aus Shenzhen und unterscheiden sich lediglich durch zwei Nukleotide. Die Genomanalyse dieser beiden Isolate ergab, dass sie nahe verwandt mit den bei Fledermäusen (englisch bat) auftretenden SARS-ähnlichen Coronaviren bat-SL-CoVZXC21 (NCBI-GenBank-Nummer MG772934) und bat-SL-CoVZC45 (NCBI-GenBank-Nummer MG772933) sind, zu letzterem besteht eine Übereinstimmung in der Nukleotidabfolge von 89 %. Das Genom der beiden Fledermaus-Coronaviren wurde 2018 sequenziert, bat-SL-CoVZC45 wurde bei der Chinesischen Hufeisennase (Rhinolophus sinicus)[62] aus der Familie der Hufeisennasen (Rhinolophidae) gefunden, die Wirtstiere wurden in Zhoushan in der ostchinesischen Provinz Zhejiang in den Jahren 2015 und 2017 untersucht.[61]
Ein weiteres Virusisolat (WIV04, NCBI-GenBank-Nummer MN996528[58]) von SARS-CoV-2 aus der bronchoalveolären Spülflüssigkeit eines der ersten Patienten zeigt ebenfalls phylogenetisch größte Ähnlichkeit mit einem bei einer anderen Fledermausart (Java-Hufeisennase, wissenschaftlich Rhinolophus affinis, englisch intermediate horseshoe bat, verbreitet in Indonesien (Java), Indien, Vietnam, China)[62] in der chinesischen Provinz Yunnan isolierten Coronavirus BatCoV RaTG13; die Genomsequenzen stimmen zu 96,2 % überein.[63][36] Auch eine am 27. Januar 2020 publizierte genetische Analyse verwies auf Fledermäuse als mutmaßlicher Ursprungswirt des Virus.[64] Am 29. Januar 2020 wurde in der Fachzeitschrift The Lancet eine genetische Analyse von zehn Virusproben publiziert, die bei neun Erkrankten gewonnen worden waren. Demnach war die Genomsequenz aller zehn Proben zu 99,98 Prozent identisch, was darauf hinweist, dass die neu entdeckte Coronavirusvariante erst vor Kurzem auf den Menschen übergegangen ist.[65][66][67] Die Genomsequenz stimmt zu 88 bzw. 87 % Prozent mit den Genomsequenzen der bei Fledermäusen auftretenden bat-SL-CoVZC45 und bat-SL-CoVZXC21 überein. Die zehn Proben zeigen hingegen nur rund 79 Prozent Übereinstimmung in der Genomsequenz zu SARS-CoV und rund 50 Prozent zu MERS-CoV. Die Ergebnisse der phylogenetischen Untersuchungen werden auch als phylogenetischer Baum, der die verwandtschaftsverhältnisse von SARS-CoV-2 innerhalb der Coronaviridae zeigt, veranschaulicht.[61][65] Eine darauf basierende Darstellung ist im Artikel Betacoronavirus zu finden.
Der Aufbau des Genoms sowohl der SARS-CoV-2-Isolate wie auch der genannten Fledermaus-Coronaviren ist typisch für Viren der Lineage B (Untergattung Sarbecovirus, englisch SARS-like Betacoronavirus) der Gattung Betacoronavirus. Aufgrund der genetischen Distanzen zu SARS-CoV und zu MERS-CoV wurde SARS-CoV-2 zunächst als eine in Bezug auf den Menschen neue, ihn infizierende Betacoronavirus-Spezies angesehen.[61][65] Aufgrund der großen genetischen Übereinstimmung mit dem ursprünglichen SARS-Coronavirus hatte am 11. Februar 2020 die Coronavirus Study Group des ICTV jedoch vorgeschlagen, das neue Virus derselben Spezies Severe acute respiratory syndrome-related coronavirus zuzuordnen wie das bisherige.[1]
Das S-Protein (S für englisch spikes) ist für die Bindung an die Wirtszelle verantwortlich, funktionell wird es in die S1-Domäne und die S2-Domäne unterschieden. Die S1-Domäne vermittelt die Bindung an den Oberflächenrezeptor der Wirtszelle, die S2-Domäne vermittelt die Fusion der Zellmembran, durch Endozytose erfolgt dann der Eintritt des Virus in die Zelle. Das S-Gen von SARS-CoV-2 zeigt mit 75 % eine eher geringe Übereinstimmung in der Nukleotidsequenz mit den beiden Fledermausisolaten bat-SL-CoVZC45 und bat-SL-CoVZXC21 im Vergleich zur Genomanalyse. Insbesondere die Nukleotidsequenz, die für die S1-Domäne codiert, unterscheidet sich von diesen deutlich (68 % Übereinstimmung) und weist aber eine größere Ähnlichkeit mit der entsprechenden Nukleotidsequenz von BatCoV RaTG13 auf. Es wurde aufgezeigt, dass SARS-CoV-2 und SARS-CoV den gleichen Zellrezeptor nutzen, das Angiotensin-konvertierende Enzym 2 (ACE2).[65] Dies konnte experimentell sicher nachwiesen werden, vergleiche COVID-19#Pathogenese.
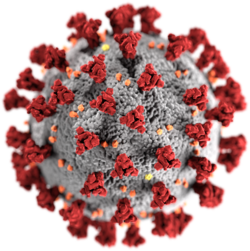
Morphologie
Die in einer Zellkultur über mehrere Tage vermehrten Viren können nach Abtrennung durch Ultrazentrifugation für die Untersuchung im Transmissionselektronenmikroskop (TEM) vorbereitet werden, dabei wird eine Negativkontrastierung verwendet. Das TEM-Bild zeigt Virionen von kugelförmiger bis pleomorpher Gestalt mit einem Durchmesser von 60 bis 140 Nanometer (nm). Auf der Oberfläche sind 9 bis 12 nm lange Spikes zu erkennen. Die Morphologie entspricht der anderer bekannter Vertreter der Familie der Coronaviridae. Die Wirtszellen, die im lichtmikroskopischen Bild einen cytopathischen Effekt aufweisen, können nach Fixierung und anschließendem Ultradünnschnitt (Dicke von 80 nm) ebenfalls mit dem TEM untersucht werden. Hier zeigen sich neben Virionen auch Einschlusskörperchen, die mit Viren gefüllte membrangebundene Vesikel im Cytoplasma enthalten.[14]
Replikationszyklus
Der Replikationszyklus der Viren verläuft in mehreren Stufen[68].
Zunächst heften sich die Virionen an die Oberfläche der Wirtszellen an. Dies geschieht spezifisch über bestimmte Oberflächenmerkmale (Rezeptoren) der Wirtszelle, im Fall von SARS-COV-2 über die Bindung des viralen Glykoprotein S an den ACE2-Rezeptor. Der ACE2-Rezeptor der Wirtszellen könnte deshalb ein möglicher Ansatzpunkt für eine Therapie sein (1).[69] Allerdings ist noch nicht ausreichend geklärt, ob weitere Zelloberflächenmoleküle als Bindungspartner für das Glykoprotein S in Frage kommen. Im Vergleich zu SARS-CoV hat das Glykoprotein S von SARS-CoV-2 eine RGD-Peptidsequenz entwickelt, womit Rezeptoren der Integrinfamilie ebenfalls als Bindungspartner in Frage kommen[70].
Nach Bindung an den ACE2-Rezeptor spaltet die membranständige Serinprotease TMPRSS2 das virale Glykoprotein S, wodurch S als fusogenes Protein aktiviert wird und der Eintritt in die Wirtszelle erfolgt. Auch TMPRSS2 ist ein potentieller Ansatzpunkt für ein wirksames Medikament (2).[71][72]
Im nächsten Schritt dringen die Erreger in die Wirtszelle ein (vereinfachte Darstellung) (3).[73]
Vor Beginn der Virusvermehrung wird die Erbsubstanz (RNA) des Virus aus dem Kapsid freigesetzt (nur ein möglicher Weg dargestellt) (4).
Nun folgt der eigentliche Vermehrungsvorgang, die Replikation. Da SARS-COV-2 über RNA positiver Polarität verfügt, kann die RNA direkt als „Bauanleitung“ (mRNA) für virusspezifische Proteine genutzt werden (Translation). Für die Wirtszelle ist die Virus-RNA praktisch nicht von eigener mRNA zu unterscheiden und der Syntheseapparat (Ribosomen) der Wirtszelle produziert virusspezifische Proteine (S, M, E, N, RNA-Polymerase) (5).[74]
Die Erbsubstanz (RNA) des Virus wird in der Wirtszelle durch Kopieren vervielfältigt (RNA-Replikation). Dazu sind die Enzyme der Wirtszelle selbst nicht in der Lage, diese Aufgabe wird von der viralen RNA-Polymerase übernommen, die viele Kopien der gesamten Virus-RNA herstellt (6).
Sind virale RNA-Kopien und Virusproteine in ausreichender Menge von der Wirtszelle hergestellt, werden sie ins endoplasmatische Retikulum (ER) aufgenommen und lagern sich zu neuen Viren zusammen (self-assembly) (7).[75]
Die fertigen Viruspartikel werden als Golgi-Vesikel aus dem ER abgeschnürt (Knospung) (8).
Durch Knospung gelangen die Viren aus der Wirtszelle (9).
Herkunft und Wirtsspektrum
Seitdem man die Krankheit als Viruskrankheit erkannt hat, werden verschiedene Tiergruppen als Ursprung oder wenigstens Überträger des Erregers diskutiert. Eine molekulare Datierungsschätzung mittels Genom-Vergleich der verschiedenen SARS-CoV-2-Isolate legt einen Ursprung der Virusvariante im November 2019 nahe.[25][39] Van Dorp und ihre Kollegen ermittelten aufgrund phylogenetischer Analysen der verschiedenen Virusvarianten anfangs Mai 2020, dass das Virus zwischen dem 6. Oktober und dem 11. Dezember 2019 auf den Menschen übergesprungen sein sollte.[54]
Schlangen und Vögel
Fachleute vermuteten zu Beginn der Epidemie in China, dass als Hauptwirt ein anderes Säugetier oder Geflügel infrage komme. Der Übergang vom Tier auf den Menschen könne jedoch über einen noch nicht identifizierten Zwischenwirt erfolgt sein.[76] Chinesische Forscher verwiesen im Journal of Medical Virology auf Schlangen wie den Vielgebänderten Krait (Bungarus multicinctus) und die Chinesische Kobra (Naja atra),[77][78] die auf dem Großmarkt, der als Infektionsort der ersten Infizierten vermutet wird, neben anderen lebenden Wildtieren (sogenannten Ye Wei) wie Fledermäusen oder Kaninchen angeboten werden.[79] Diese Hypothese wurde von anderen Virologen für unwahrscheinlich erklärt,[80] da es bisher keine Evidenz dafür gebe, dass Coronaviren auch Reptilien infizieren können. Bisher seien Coronaviren ausschließlich bei Säugetieren und Vögeln gefunden worden.[76]
Schuppentiere und Fledermäuse
Hufeisennasen-Fledermäuse – möglicherweise mehrere höhlenbewohnende Arten – waren das Reservoir des Erregers SARS-CoV[-1] das die SARS-Epidemie einige Jahre zuvor ausgelöst hatte, mit dem Larvenroller (Paguma larvata) als möglichem Zwischenwirt zwischen Fledermaus und Mensch. Seitdem wurden verschiedene weitere Betacoronaviren (insbesondere auch SARS-artige der Untergattung Sarbecovirus) vor allem bei Fledermäusen, aber auch bei Menschen gefunden.[25]
SARS-ähnliche Coronaviren (d. h. der Untergattung Sarbecovirus) wurden 2017 in Höhlen in der chinesischen Provinz Yunnan in verschiedenen Hufeisennasenarten gefunden: der Java-Hufeisennase (Rhinolophus affinis, englisch intermediate horseshoe bat), der Chinesischen Hufeisennase (R. sinicus) und der Großen Hufeisennase (R. ferrumequinum); sowie in der Stoliczka-Dreizackblattnase (Aselliscus stoliczkanus).[37]
Aufgrund der Ähnlichkeit der Bindungsstelle (englisch receptor binding domain, RBD) des Spike-Proteins an den menschlichen Rezeptor ACE2 gilt inzwischen das Virus-Isolat BatCoV RaTG13[81] (gefunden in Java-Hufeisennasen Rhinolophus affinis, englisch intermediate horseshoe bat in Yunnan), als wichtiger Kandidat für den Ursprung von SARS-CoV-2, auch wenn nicht klar ist, ob die Übertragung direkt erfolgte. Die Übereinstimmungen der Genom-Sequenz zwischen RaTG13 und SARS-CoV-2 beträgt 96 %.[36][25]
Nachdem in Malaiischen Schuppentieren (Manis javanica, englisch Sunda pangolin) Coronaviren mit hoher genetischer Übereinstimmung zum SARS-CoV-2 gefunden wurden (Manis-CoV,[80] Isolate SRR10168377 und SRR10168378),[39] gerieten diese in Verdacht, der Ursprung der Pandemie zu sein, zumal diese trotz Verbots in China gehandelt werden.[34][36][82][83][84][85][80] Die Übereinstimmung betrug in diesem Fall 90 % über das gesamte Genom, aber 99 % in einer spezifischen Region des Spike-Proteins (S-Protein), die es dem Virus erlaubt, an die ACE-Rezeptoren der menschlichen Zellen zu binden.[25]
Interessanterweise ist das in den Java-Hufeisennasen R. affinis isolierte Virus RaTG13 gerade in diesem Genom-Abschnitt zu SARS-CoV-2 mit nur 77 % Übereinstimmung vergleichsweise unterschiedlich.[25]
Dies bedeutet, dass die aus den Malaiischen Schuppentieren isolierten Coronaviren in menschliche Zellen eindringen können, das aus Java-Hufeisennasen isolierte jedoch nicht.[25] Außerdem ist dieses Ergebnis verträglich mit der Annahme, dass das neuartige Coronavirus SARS-CoV-2 das Ergebnis einer Rekombination der RNA-Moleküle zweier verschiedener Viren sein könnte, eines dem RaTG13 aus Fledermäusen, das andere den beiden Isolaten aus den Schuppentieren nahestehend. Dann wäre SARS-CoV-2 eine neue Chimäre aus diesen beiden früheren Viren.[25][86]
Zwar besitzen Coronaviren anders als etwa Influenzaviren ein unsegmentiertes Genom (monopartit), d. h. nur ein einziges Nukleinsäuremolekül (hier RNA). Dass es dennoch auch bei dieser Virusfamilie einen Rekombinationsmechanismus gibt, wurde bereits früher beschrieben, insbesondere um den Ursprung des alten SARS-Virus SARS-CoV[-1] zu erklären.[25][87] Eine solche Rekombination kann, egal ob segmentiertes oder unsegmentiertes Genom, zu einem neuen Virus führen, das eine neue Wirtsspezies befallen und krank manchen kann.[25] Das Rekombinationsereignis kann daher zum Ausgangspunkt einer neuen Epidemie werden, wie es bei SARS vermutet (und bei Influenza stets befürchtet) wird. Voraussetzung ist die Doppelinfektion (Koinfektion) eines (einzelnen) Wirtsindividuums durch die beiden Ausgangsviren.[25]
Allerdings bleibt bislang (Stand 25. März 2020) ungeklärt, in welcher Spezies die hypothetische Doppelinfektion stattgefunden haben könnte, und unter welchen Umständen dies geschehen sein könnte.[25]
Marderhunde als mögliche Zwischenwirte
Laut Christian Drosten könnten Marderhunde (Nyctereutes procyonoides) möglicherweise die gesuchten Zwischenwirte sein. Auch das ursprüngliche SARS-Virus (SARS-CoV-1) wurde in Marderhunden gefunden, die wegen ihres Fells in China gezüchtet werden und somit als Überträger auf den Menschen in Frage kommen.[88][89][90]
Haustiere als Wirte
Laut WHO gibt es keine Hinweise, dass Haustiere SARS-CoV-2 als Träger weiterverbreiten (Stand: Anfang März).[91][92] Jedoch können einige andere Viren aus der Corona-Virusfamilie Coronaviridae auch bei Haustieren Erkrankungen auslösen, z. B. die beiden Alphacoronaviren CCoV (Hunde) und FCoV (Katzen).[93]
Am 28. Februar 2020 gab die Regierung Hongkongs bekannt, erstmals einen Hund positiv auf das Virus getestet zu haben, der im Haushalt seiner infizierten Halter lebte.[94] Die WHO bestätigte, die SARS-CoV-2-Proben seien „schwach positiv“[95] getestet worden. Obwohl bei dem Hund das Virus im Blut nachgewiesen werden konnte,[96] löste es bei diesem keine klinisch nachweisbaren Hinweise auf eine Erkrankung aus.[92] Das Tier wurde zuletzt am 12. und 13. März 2020 mit negativem Befund auf SARS-CoV-2 getestet, so dass seine Quarantäne beendet und es dem Besitzer zurückgegeben wurde. Zwei Tage nach Ende der Quarantäne verstarb der Hund, ohne dass ein direkter Zusammenhang mit dem Virusbefall nachweisbar war.[97]
Mitte März wurden in Hongkong zwei weitere Hunde positiv auf SARS-CoV-2 getestet, die ebenfalls ohne auffällige Symptome einer Infektion waren.[98]
In Lüttich (Belgien) wurde Ende März 2020 die Hauskatze eines Infizierten positiv auf SARS-CoV-2 getestet.[99] Das Tier erkrankte an Durchfall, hatte Atemprobleme und verstarb.[100] Eine Ende März 2020 in Hongkong bei einer Hauskatze nachgewiesene Infektion verlief hingegen symptomlos.[101] Antikörpernachweise hatten zuvor bereits in Wuhan ergeben, dass dort auch Katzen infiziert worden waren.[102] Zudem wurde mehrfach in Laborexperimenten belegt, dass infizierte Katzen die Viren an andere Katzen weitergeben können.[103][104]
Mitte April 2020 erschien ein Artikel über die Möglichkeit von streunenden Hunden als Zwischenwirt für die Übertragung von Sarbecoviren (RaTG13, Pangolin-CoV) von Wildtieren (Fledermäusen, Schuppentieren) auf den Menschen. Eine wichtige Rolle spielt dabei das Zinkfingerprotein ZAP.[105][106]
Weitere Wirbeltiere
Im New Yorker Bronx Zoo wurde Anfang April 2020 ein erwachsener Tiger positiv auf SARS-CoV-2 getestet,[107] nachdem bei ihm trockener Husten und keuchender Atem aufgefallen waren, jedoch keine Atemnot. Weiterhin wiesen auch zwei Löwen und fünf Tiger ähnliche Symptome auf, weswegen auch bei ihnen eine Infektion mit SARS-CoV-2 vermutet wurde. Infiziert wurden die Tiere vermutlich von einem asymptomatischen Bediensteten des Zoos. Wenige Tage nach dem Auftreten von Krankheitszeichen erholten sich die Tiere wieder.[108]
Durch Laborexperimente in Korea wurde belegt, dass Frettchen empfänglich für eine SARS-CoV-2-Infektion sind und diese auch an Artgenossen weitergeben können.[109] Das Friedrich-Loeffler-Institut bestätigte aufgrund eigener Test den Befund aus Korea und wies zugleich darauf hin, dass auch Nilflughunde empfänglich für eine SARS-CoV-2-Infektion sind, Schweine und Hühner hingegen nicht. Insbesondere die Empfänglichkeit von Frettchen sei ein wichtiger Befund, „da sie als Modelltiere für die Infektion des Menschen zur Erprobung von Impfstoffen oder Medikamenten eingesetzt werden könnten“.[110] Chinesische Forscher berichteten im April 2020 in der Fachzeitschrift Science, dass sich das Virus in Hunden, Schweinen, Hühnern und Enten nur schlecht („poorly“) vermehre, und bestätigten, dass Frettchen und Katzen infiziert werden können.[111] Auch Goldhamster, die nach einer Infektion mit SARS-CoV[-1] nur sehr schwache Symptome entwickelt hatten und daher als Modelltiere ungeeignet waren, ließen sich im Labor mit SARS-CoV-2 infizieren, zeigten deutliche Symptome und wiesen hohe Viruskonzentrationen in Lunge und Darm auf.[112]
Im April und Mai 2020 wurden Infektionen und Erkrankungen in vier niederländischen Nerzfarmen festgestellt. Das Virus wurde von infizierten Mitarbeitern eingeschleppt und vermutlich auch zwischen den Tieren weitergegeben. Die erkrankten Nerze zeigen ähnliche Symptome wie Menschen: Atemwegsbeschwerden, Probleme mit dem Verdauungstrakt, erhöhte Sterblichkeit.[113]
Primaten
Ob neben dem Menschen noch Menschenaffen an SARS-CoV-2 erkranken können, ist derzeit (Stand Ende März 2020) noch ungeklärt.[114]
- Im Jahr 2016 wurde bei Schimpansen im Tai-Nationalpark (Elfenbeinküste) eine Infektion mit dem Humanen Coronavirus OC43 (HCoV-OC43, ein Betacoronavirus aus der Untergattung Embecovirus, Spezies Betacoronavirus 1)[115] beobachtet, das bei Menschen milde erkältungsartige Symptome hervorruft. Diese zeigten auch die Schimpansen. Es ist daher nicht ausgeschlossen, dass auch SARS-CoV-2 vom Menschen zumindest auf Menschenaffen überspringen könnte. Es wurde (insbesondere für Wildhüter) empfohlen, zu den Wildtieren einen Mindestabstand von 7 bis 10 Meter zu halten und auch gegenüber den Tieren entsprechende Quarantänezeiten einzuhalten.[116]
- Neben Schimpansen (einschließlich Bonobos) könnten auch Gorillas und Orang-Utans bedroht sein, so wie die Schimpansen und Gorillas vom Humanen Metapneumovirus (HMPV, Familie Pneumoviridae).[117][116]
- Eine chinesische Forschergruppe um Chuan Qin stellte im März 2020 vorläufige Ergebnisse ihrer Untersuchungen an Rhesusaffen als Preprint zur Verfügung. Hierbei ging es insbesondere um die Frage der Infektiosität nach überstandener Erkrankung.[118][119][120] Auch eine im Mai 2020 in Science publizierte Studie an Rhesusaffen berichtete von „schützender Immunität“ nach erstmaliger Erkrankung.[121]
- Niederländische Forscher berichteten im März 2020 in Science, dass SARS-CoV-2 bei Javaneraffen eine „COVID-19-ähnliche Krankheit“ verursache, weswegen diese Tiere als Modell für das Testen von vorbeugenden und therapeutischen Strategien geeignet seien.[122]
Risikogruppe nach Biostoffverordnung
Für Beschäftigte, die durch ihre berufliche Tätigkeit mit Infektionserregern in Kontakt kommen können, gilt in Deutschland die Biostoffverordnung (BioStoffV). Der bei der Bundesanstalt für Arbeitsschutz und Arbeitsmedizin (BAuA) eingerichtete Ausschuss für Biologische Arbeitsstoffe (ABAS) hat SARS-CoV-2 am 19. Februar 2020 vorläufig in die Risikogruppe 3 nach der BioStoffV eingeordnet (zweithöchste Stufe).[123] Grundsätzlich erfolgt die Einstufung in Risikogruppen in den Technischen Regeln für biologische Arbeitsstoffe (TRBA), die von der BAuA veröffentlicht werden, für Viren ist dies die TRBA 462: Einstufung von Viren in Risikogruppen. Beim Auftreten neuartiger, noch nicht zugeordneter Krankheitserreger erfolgt zunächst eine vorläufige Einstufung durch den ABAS. In der Begründung wird auf die Ähnlichkeit von SARS-CoV-2 mit dem SARS-CoV-1 hingewiesen, der die SARS-Pandemie 2002/2003 ausgelöst hat, und auch die Ähnlichkeit in geringerem Umfang mit dem MERS-CoV wird erwähnt. Diese beiden Viren wurden ebenfalls der Risikogruppe 3 zugeordnet. Der ABAS nennt die „derzeit fehlenden Möglichkeiten zu Impfprävention und Therapie sowie die große Verbreitungsmöglichkeit in der Bevölkerung“ als Begründung für die vorläufige Zuordnung zur Risikogruppe 3.[124]
Außerdem werden Empfehlungen zur Arbeit mit dem Virus bei der Diagnostik im Labor gegeben: Nicht gezielte Tätigkeiten (vergleiche § 5 BioStoffV) – ausgehend vom Untersuchungsmaterial, also beispielsweise die Probenvorbereitung, Probenaufbereitung und die Inaktivierung, um den Nachweis mittels RT-PCR (siehe Abschnitt Nachweismethoden) durchzuführen – können unter den Bedingungen der Schutzstufe 2 durchgeführt werden. Dabei sind alle Tätigkeiten, bei denen mit Aerosolbildung zu rechnen ist, in einer mikrobiologischen Sicherheitswerkbank der Klasse II durchzuführen. Außerdem ist die entsprechende persönliche Schutzausrüstung zu tragen. Gezielte Tätigkeiten nach § 5 BioStoffV dürfen nur in Laboratorien der Schutzstufe 3 durchgeführt werden, dies betrifft z. B. die Vermehrung des Virus in einer Zellkultur.[124] Die amerikanische Gesundheitsbehörde CDC hatte zuvor ähnliche Empfehlungen herausgegeben.[125]
Krankheitsentstehung
Durch das Virus SARS-CoV-2 wird die Krankheit COVID-19 (für englisch coronavirus disease 2019 ‚Coronavirus-Krankheit 2019‘) ausgelöst. Informationen zu Übertragungsweg, Inkubationszeit, Symptomen und Behandlung finden sich im diesbezüglichen Artikel. Die weltweite Ausbreitung der Krankheit ist im Artikel COVID-19-Pandemie beschrieben.
Klinische Erscheinungen
| Klassifikation nach ICD-10 | |
|---|---|
| U07.1 | COVID-19, Virus nachgewiesen |
| U07.2 | COVID-19, Virus nicht nachgewiesen |
| ICD-10 online (WHO-Version 2019) | |
Nachweismethoden
Vorgehensweise beim Nachweis
- → Siehe: COVID-19#Diagnostik
RT-PCR
Die Nachweismethode der Charité ist die real-time quantitative Reverse-Transkriptase-Polymerase-Kettenreaktion (abgekürzt als qRT-PCR oder RT-qPCR).
Dabei reagiert der Test auf das Vorhandensein zweier bestimmter kurzer Gensequenzen (Nukleotidsequenzen), die kennzeichnend für die genannten Viren sind, bezeichnet als E-Gen und RdRp-Gen. Das E-Gen codiert für die Virushülle (E für engl. envelope ‚Hülle‘), das RdRp-Gen für die RNA-abhängige RNA-Polymerase (RdRp für engl. RNA-dependent RNA polymerase). Der Test reagiert auch auf bloße Virenreste und würde auch alle anderen Keime und Lebensformen erkennen, die (jetzt oder in Zukunft) zufällig in diesen genetischen Kennzeichen mit SARS-CoV-2 übereinstimmen.[23][126][127]
Ablauf
Der Test beinhaltet drei standardisierte Einzeltests (im Dt. „Assays“ genannt, aus dem Engl.) in einem definierten Verfahrensablauf:
- Screening: Das erste Assay (E-Gen) dient als Screening, da es mehrere Virusspezies der Untergattung Sarbecovirus (aus der Gattung Betacoronavirus) nachweist.
- Bestätigung: Verläuft das Screening positiv, so ist ein Bestätigungs-Assay (RdRp-Gen) durchzuführen.
- Charakterisierung: Falls auch dort ein positives Ergebnis erhalten wird, schließt sich als drittes ein Charakterisierungs-Assay an (ebenfalls RdRp-Gen).
Die letzten beiden Assays verwenden (neben anderen) eine Gensonde, die für SARS-CoV-2 spezifisch ist. Beim zweiten Assay ist zusätzlich eine Sonde enthalten, die zur Nukleotidsequenz sowohl des RNA-Teilstücks des SARS-CoV-1 als auch des SARS-CoV-2 passt.[23][127]
Für die Durchführung der qRT-PCR werden 90 Minuten bis drei Stunden benötigt.[128] Anstelle der real-time quantitative-Variante können die Produkte der Reverse-Transkriptase-PCR auch mittels Agarose-Gelelektrophorese nachgewiesen werden.[61]
Entwicklung
Die erste Version des real-time RT-PCR-Assays ist erstellt worden, bevor die Genomsequenz von SARS-CoV-2 veröffentlicht war. Für das Primerdesign wurden hier noch das erste SARS-assoziierte Coronavirus SARS-CoV-1 und weitere, bei Fledermausarten vorkommende SARS-assoziierte Coronaviren (Sarbecoviren[129]) verwendet. Nach der Veröffentlichung der Genomsequenz wurden die Primer ausgewählt, die für den Nachweis von SARS-CoV-2 geeignet sind.[23][127]
Die molekularbiologische Nachweismethode, die im Konsiliarlabor an der Charité verwendet wird, wurde in Zusammenarbeit mit der Berliner Biotechnologiefirma TIB Molbiol im Schnellverfahren entwickelt,[130] eine erste Version war bereits am 13. Januar 2020 verfügbar.[127] Dazu haben chinesische Wissenschaftler bereitwillig unveröffentlichte Befunde beigetragen. Von internationalen Forschungsnetzen kamen grundlegende Daten, und die globale Sektion des Europäischen Virus-Archivs (EVAg) lieferte notwendige Produkte (SARS-CoV-1-RNA und RNA-Transkripte) für die Assays.[127] Auch weitere Gruppen von Wissenschaftlern haben ihre entwickelten Methoden veröffentlicht. Dabei handelt es sich um PCR-Protokolle oder Auflistungen geeigneter Primer und deren für die RT-PCR verwendete Stoffmengenkonzentration, beispielsweise von den Centers for Disease Control and Prevention (CDC) in den USA, den CDC in China oder der Universität Hongkong. Sie unterscheiden sich darin, welche Gene der Virus-RNA nachgewiesen werden, beispielsweise das N-Gen (N für das Nukleokapsid-Phosphoprotein), das ORF1ab-Gen (codiert für das ORF1ab-Polyprotein) oder das Gen für das Spike-Protein.[131] Von der in Berlin entwickelten Nachweismethode wurden in den ersten zwei Monaten bereits 40.000 Testkits, zur Untersuchung von 4 Millionen Proben, in über 60 Länder verkauft.[132]
In Deutschland wurde im März 2020 eine Methode vorgestellt, bei der Proben mehrerer Testpersonen zusammengeführt und gemeinsam getestet werden. Ein negativer Befund bedeutet, dass die Ergebnisse aller Proben zuverlässig negativ sind, und nur bei einem positiven Befund müssen die Proben einzeln untersucht werden. Daher lassen sich, so die Forscher, mit vergleichbarem Aufwand deutlich mehr Personen testen als mit herkömmlichen Verfahren, sofern die untersuchten Personen eine geringe Infektionswahrscheinlichkeit aufweisen. Damit ließe sich, so die Forscher, in Deutschland die Zahl der täglichen Untersuchungen auf 200.000 bis 400.000 steigern.[133]
Aussagekraft
Die wichtigsten Gütekriterien für die Aussagekraft (Validität) diagnostischer Labortests sind Spezifität und Sensitivität. Unter Spezifität wird die Wahrscheinlichkeit verstanden, dass eine eigentlich negative Probe auch als negativ erkannt wird (Ausschluss von Falsch-Positiven). Unter Sensitivität dagegen, die Wahrscheinlichkeit, dass eine eigentlich positive Probe auch als positiv erkannt wird (Ausschluss von Falsch-Negativen).
Spezifität
Dieser Test reagiert positiv auf SARS-CoV-2 und das SARS-CoV-1. Die Kreuzreaktivität auf SARS-CoV-1 ist gewollt, um SARS-CoV-1 (aus Laborbeständen) als positive Testkontrolle verfügbar zu machen.[134] Weitere Daten aus der Test-Entwicklung deuten darauf hin, dass der Test auf „wahrscheinlich alle asiatischen Viren“[135] (übersetzt aus dem Englischen) aus der Untergattung Sarbecovirus[129] ein positives Ergebnis liefert.
In der Entwicklung wurde sichergestellt, dass der Test nicht positiv auf die endemischen humanen Coronaviren (HCoV-229E, -NL63, -OC43, -HKU1), das MERS-CoV und viele andere übliche Erreger von respiratorischen Erkrankungen reagiert. Wie andere etablierte Teste für menschliche Coronaviren reagiert auch dieser Test positiv auf verschiedene, beim Menschen unbekannte Coronaviren (insbesondere solche von bestimmten Fledermausarten).[126]
Um festzustellen, dass der Test nicht auf ein anderes Virus ungewollt positiv reagiert, werden Proben getestet, die das Zielvirus nicht enthalten. Somit wird hier sichergestellt, dass der Test eine negative Probe in diesen Fällen auch tatsächlich als negativ anzeigt. Aufgrund dieser Untersuchungen wird die Spezifität dieses Tests als äußerst hoch eingeschätzt, sofern (aus anderen Erwägungen) sichergestellt werden kann, dass die getestete Probe frei von SARS-CoV-1 und anderen asiatischen Sarbecoviren ist.[126]
Dass dennoch selbst lange etablierte Teste im Nachhinein Mängel bei der Spezifität zeigen können, zeigt ein Beispiel aus dem Jahre 2006 eines PCR-Tests für das dem SARS-CoV-2 nahe verwandte SARS-CoV-1. Es zeigte sich, dass dieser SARS-CoV-1-Test auch positiv auf das Gattungs-verwandte humane Betacoronavirus HCoV-OC43 reagierte. Hier hatten auch parallele Antikörperteste das falsch-positive Ergebnis zunächst untermauert.[136] Daraus ergibt sich, dass diese Kreuzreaktion auch bei anderen SARS-CoV-1-Testen vorkommen könnte.
Obwohl der in diesem Abschnitt beschriebene SARS-CoV-2-Test ebenfalls gleichzeitig einen SARS-CoV-1-Test darstellt, ist diese spezielle Kreuzreaktion hier jedoch evident ausgeschlossen, da er, wie oben erwähnt, speziell auch gegen das HCoV-OC43 validiert wurde. Mutationen am HCoV-OC43 könnten das aber in Zukunft auch wieder ändern.
Sensitivität
Während die hohe Spezifizität weitgehend akzeptiert wurde, wird die Sensitivität des Tests des öfteren kritisiert und es wird von häufigen falsch-negativen Ergebnissen berichtet. Im Ergebnis führte das z. B. bei mehrfach hintereinander getesteten Patienten dazu, dass der Status immer wieder zwischen positiv, negativ und unklarem Ergebnis wechselte. Das Problem wird hier aber nicht auf der „technischen“ Seite des Tests gesehen, sondern in der richtigen Ausführung und Handhabung.[137][138]
Womöglich werden zu wenige Proben, Proben von den falschen Stellen oder Proben auf die falsche Art entnommen. Das kann dazu führen, dass das Virus in der Probe fehlt, aber im Menschen trotzdem vorhanden ist. Der Test fällt dann „technisch-korrekt“ negativ aus, obschon der Mensch Spuren des Virus in sich trägt.[139][137][138]
Weitere Methoden
Genomanalyse
Laboratorien mit Ausstattung für eine Genomanalyse (DNA-Sequenzierung des Genoms), also einem Sequenzierautomaten, können SARS-CoV-2 auch auf diese Weise identifizieren.[140] Vollständige Genomanalysen von SARS-CoV-2-Isolaten zum Vergleich sind beispielsweise in der Gendatenbank des National Center for Biotechnology Information (NCBI) oder über die GISAID Plattform[141] verfügbar (vergleiche Abschnitt Molekulargenetik).
Nukleinsäurenachweis

Die Weltgesundheitsorganisation (WHO) berichtete bereits Mitte Januar 2020 über die Entwicklung von vereinfachten molekularbiologischen Verfahren, die Nucleic Acid Amplification Technology (NAAT), deren Assays validiert wurden. Die NAAT-Methode beruht ebenfalls auf der RT-PCR, das fertig zusammengestellte Assay bietet jedoch den Vorteil, einfacher in der Handhabung zu sein und lässt sich von entsprechend ausgestatteten Routine-Laboratorien verwenden.[140]
Am 5. Februar 2020 gab die US-amerikanische Behörde CDC bekannt, ein derartiges Assay (Testkit) für die Anwendung in akkreditierten Diagnoselaboratorien zur Verfügung zu stellen. Das Assay wird als Centers for Disease Control and Prevention (CDC) 2019-Novel Coronavirus (2019-nCoV) Real-Time Reverse Transcriptase (RT)-PCR Diagnostic Panel bezeichnet und ist für den Nachweis sowohl des neuartigen Coronavirus wie auch SARS-ähnlicher Coronaviren in Proben der oberen und unteren Atemwege von Patienten vorgesehen. Zuvor erfolgte eine beschleunigte Zulassung durch die Gesundheitsbehörde Food and Drug Administration (FDA), somit darf das Assay seit 4. Februar 2020 auch außerhalb von Forschungseinrichtungen verwendet werden.[142] Ein Testkit ermöglicht die Untersuchung von 700 bis 800 Proben, 100 dieser Packungen gingen an US-amerikanische Labore, weitere 100 an internationale Laboratorien, die beispielsweise im Auftrag der WHO Untersuchungen durchführen.[143] Die Untersuchung dauert von der Probevorbereitung bis zum Vorliegen der Ergebnisse etwa vier Stunden.[128] Das Hamburger Unternehmen Altona Diagnostics sowie das Darmstädter Unternehmen R-Biopharm bieten seit Februar 2020 ein ähnliches Testkit für die RT-PCR an, das ebenfalls weltweit vertrieben wird. Ein Testkit ermöglicht die Analyse von etwa 100 Proben.[144] Allerdings sind diese bisher nur für die Verwendung in der Forschung zugelassen.[145]
Schnelltest
Der in den Medien verwendete Begriff Schnelltest ist nicht klar definiert, einerseits ist damit die Erwartung verbunden, die Zeitdauer bis zum Vorliegen eines Testergebnisses im Vergleich zur mittlerweile routinemäßig eingesetzten RT-qPCR-Methode zu verkürzen,[146] andererseits kann damit auch gemeint sein, dass es sich um ein Point-of-Care-Testing (deutsch patientennahe Labordiagnostik) handelt,[146] bei dem der Schritt „Probentransport zur Analyse im Zentrallabor“ entfällt, da entsprechende Geräte vor Ort, beispielsweise in einer Arztpraxis verwendet werden. Somit werden auch Kartuschensysteme, die auf der RT-PCR-Methode beruhen, in den Medien manchmal als Schnelltest bezeichnet, hier aber in einem eigenen Abschnitt dargestellt.
Ende Januar 2020 hatte Xinhua berichtet, dass die chinesische Behörde National Medical Products Administration (NMPA) am 26. Januar vier Testkits eines neuen Testverfahrens zugelassen habe. Das Assay ist von dem Biotechnologieunternehmen Sansure Biotech aus Changsha entwickelt worden. Mit Hilfe der dafür geeigneten Laborautomatisierung sollen Testergebnisse bereits nach 30 Minuten vorliegen.[128][147] Die staatliche Nachrichtenagentur teilte weiterhin mit, dass eine in Wuxi in der östlichen Provinz Jiangsu ansässige Firma in Zusammenarbeit mit dem National Institute for Viral Disease Control and Prevention eine Schnellmethode entwickelt habe. Mit dem Testkit soll das Virus innerhalb von 8 – 15 Minuten nachgewiesen werden, die Firma könne täglich so viele Testkits produzieren, dass damit die Untersuchung von 4000 Proben möglich sei und das Verfahren soll bereits in der Provinz Hubei eingesetzt worden sein.[148] Die Xinhua-Meldungen enthalten keinen Hinweis auf die dabei verwendeten molekularbiologischen Methoden. Weiterhin wird an der chinesischen Tianjin-Universität in Zusammenarbeit mit einer Pekinger Biotechnologiefirma ein Schnellverfahren entwickelt, bei dem nach 15 Minuten Resultate vorliegen sollen. Es befindet sich im Probeeinsatz (Stand Februar 2020).[128]
Forscher der Hong Kong University of Science and Technology meldeten Anfang Februar 2020 die Entwicklung eines tragbaren Gerätes, mit dem das neuartige Coronavirus innerhalb von 40 Minuten nachweisbar sein soll. Für das im Vergleich zur herkömmlichen qRT-PCR schnellere Verfahren werden modifizierte Chip-Thermocycler verwendet.[128] Auch Forscher des Institute for Health Innovation & Technology (iHealthtech) an der National University of Singapore berichteten im Februar 2020 darüber, eine Schnellmethode zu entwickeln. Sie basiert auf der seit 2018 verwendeten enVision-Technologie, mit der Nukleinsäuren innerhalb von 30 bis 60 Minuten nachgewiesen werden. Es wird geschätzt, dass bis zur Marktreife des neuen Testverfahrens noch mehrere Monate benötigt werden.[149]
Eine deutsche Forschergruppe der Universität Bonn um Hendrick Streeck hat im April 2020 die Ergebnisse einer Schnelltestvalidierung vorgestellt[150]. Hierbei wurde der CoV-2 Rapid Test der Firma PharmACT im Rahmen eines Screenings der Bevölkerung getestet und mit parallel gewonnenen Proben zur PCR-Diagnostik verglichen. Von insgesamt 49 Personen waren 22 positiv in der PCR; der Schnelltest erkannte jedoch nur acht davon richtig als positiv (Sensitivität 36,4 %). Von den 27 PCR-negativen Personen wurden durch den Schnelltest nur 24 richtig als negativ diagnostiziert (Spezifität 89,9 %).
Kartuschentest
Bei einem Kartuschensystem wird ein technisch aufwändiges, aber dennoch von den Größenabmessungen transportables Gerät (engl. analyzer) verwendet, in dem eine dafür konstruierte Kartusche (engl. cartridge) eingesetzt wird. Die Kartusche wird zuvor mit dem Probenmaterial, z. B. dem Abstrichtupfer bestückt, weitere Chemikalien und biologische Arbeitsstoffe für die Probevorbereitung und die Analyse sind in der Kartusche enthalten. Die für den Einmalgebrauch konzipierte Kartusche verkürzt die Dauer bis zum Vorliegen des Testergebnisses und bietet für den Benutzer den Vorteil, den Kontakt mit den Infektionserregern zu minimieren (vergleiche Abschnitt Risikogruppe nach Biostoffverordnung). Kartuschentests, auch als engl. panel bezeichnet, werden seit 2018 für die Diagnostik von Krankheitserregern, die Atemwegserkrankungen verursachen, eingesetzt. Die Methode basiert auch hier auf der RT-qPCR, der real-time quantitativen Reverse-Transkriptase-Polymerase-Kettenreaktion, da aber gleichzeitig Gene mehrerer Krankheitserreger analysiert werden, wird sie der Multiplex-PCR zugerechnet.[146]
Das Biotechnologieunternehmen Qiagen N.V. entwickelte am Standort in Hilden einen Kartuschentest als Schnelltest, der auf einem bereits international für die Diagnostik von Krankheitserregern zugelassenen Verfahren basiert, mit dem sich u. a. SARS-assoziierte Viren und EHEC nachweisen lassen.[151] Das Verfahren wurde um den Nachweis der im SARS-CoV-2-Genom vorhandenen Gene ORF1b und E erweitert und die Ergebnisse wurden mit denen der RT-PCR-Methode verglichen.[152] Das Unternehmen arbeitete mit der WHO zusammen, um eine Validierung zu erreichen.[151] Das tragbare Diagnosegerät ist für den Einsatz in Arztpraxen oder an Flughäfen geeignet. Als Probenmaterial ist ein Abstrich aus dem Rachenraum oder eine Blutprobe geeignet,[151] Testergebnisse liegen innerhalb von 60 Minuten vor.[152] In Deutschland ist die vorläufige Zulassung durch das Bundesinstitut für Arzneimittel und Medizinprodukte beantragt.[153] Die Diagnosegeräte wurden im Februar 2020 in französischen und chinesischen Krankenhäusern getestet[152] und erhielten die Zulassung der US-amerikanischen und europäischen Behörden (Stand 27. März 2020).[154] Im Vergleich zur routinemäßig eingesetzten RT-qPCR-Methode sind Kartuschentests teurer, zudem wird das dazugehörige Analyse-Gerät benötigt.[154]
Zwei weitere Diagnostik-Firmen in Deutschland entwickeln ebenfalls derartige Schnelltests.[153] Die US-amerikanische Firma Cepheid Inc hat im März 2020 eine beschleunigte Zulassung durch die FDA erhalten. Auch hier handelt es sich um ein Kartuschensystem auf der Basis von RT-qPCR mit dazugehörigem Analyzer, Testergebnisse sollen nach 45 Minuten vorliegen.[154] Von den passenden Analyse-Geräten werden zur Zeit weltweit etwa 23.000 verwendet, zumeist in Krankenhäusern, ein Analyzer kann bis zu vier Kartuschen gleichzeitig aufnehmen (Stand 27. März 2020).[154] Die Robert Bosch GmbH entwickelte zusammen mit der britischen Firma Randox Laboratories ebenfalls einen Kartuschentest, mit dem Testergebnisse nach 2,5 Stunden vorliegen sollen. Das Testsystem soll im April 2020 auf den Markt kommen, hat bisher jedoch nur eine Zulassung für Forschungseinrichtungen.[154]
Antikörpernachweis
Die Weltgesundheitsorganisation (WHO) berichtete Mitte Januar 2020 über die Entwicklung von Antikörpernachweisen als serologische Untersuchung.[140] Dadurch wird es ein Assay geben, beispielsweise ein Immunassay wie ELISA oder einen Lateral Flow Test, mit dem Antikörper aus Patientenproben (Blutserum) durch Antigen-Antikörper-Reaktion nachweisbar sind. Der Lateral Flow Test kann als Schnelltest (siehe oben) bezeichnet werden, im Sinne von Point-of-Care-Testing (deutsch: patientennahe Labordiagnostik), ein bekanntes darauf basierendes Beispiel ist der Schwangerschaftstest.
Je nachdem, auf welche Antikörper geprüft wird, ist noch eine aktuelle Infektion nachweisbar (frühe Antikörper Immunglobulin M (IgM)) oder eine bereits abgeschlossene Infektion durch späte Antikörper Immunglobulin G (IgG),[155] dieser Antikörperklassenwechsel wird als Serokonversion bezeichnet. Es gab den Vorschlag, IgM-Antikörpernachweise zur Diagnose akuter Infektionen zu nutzen,[155] allerdings zeigte die Untersuchung chinesischer Wissenschaftler von 535 Plasmaproben von 173 Patienten, dass im Zeitraum 1 bis 7 Tage nach Einsetzen der Symptome nur bei knapp 30 % der Patienten IgM nachweisbar waren, während es im Zeitraum 8 bis 14 Tage 73 % der Patienten waren. Damit kann der IgM-Antikörpernachweis die PCR-Testung nur ergänzen.[156]
Die WHO und das Robert Koch-Institut in Deutschland rufen dazu auf, Serumproben von bestätigten oder Verdachtsfällen in der Akutphase zu sammeln und zu asservieren.[157] Die WHO empfiehlt, die erste Probe in der ersten Krankheitswoche und die zweite Probe drei bis vier Wochen später zu nehmen. Damit lässt sich eine Serokonversion überprüfen.[140][158] Nach Kontakt mit SARS-CoV-2 werden nach etwa drei Wochen (entspricht etwa zwei Wochen nach Auftreten der Symptome) die Antikörper Immunglobulin A (IgA) gebildet, nach etwa 4 Wochen die IgG-Antikörper.[159] Bei der Untersuchung von 153 Patienten wurde ermittelt, dass die Serokonversion 20–30 Tage nach Einsetzen der Symptome erfolgt, also IgG-Antikörper in ausreichender Menge vorhanden sind.[156] Antikörpertests auf IgA und IgG sind somit nicht für die Akutdiagnostik erkrankter Patienten gedacht und ersetzen nicht die PCR-Analytik.[159] Ihre Ergebnisse liefern epidemiologische Daten, mit denen das Ausmaß des Ausbruchs ermittelt werden kann und helfen bei der Überprüfung der Wirksamkeit von Impfstoffen.[140][160]
Für die Aussagekraft des Antikörpertests ist die Wahl des richtigen Antigens ausschlaggebend. Falls ein bestimmter Antikörper an mehr als einem Antigen bindet, handelt es sich um eine Kreuzreaktivität, die zu falsch positiven Testergebnissen führt, da mehr als das eigentliche Antigen reagiert.[161] Als Antigene von SARS-CoV-2 sind die im Abschnitt Merkmale beschriebenen Strukturen geeignet, beispielsweise das Nukleokapsidprotein (N) oder das Spikeprotein (S) als Ganzes bzw. alternativ die S1- und S2-Domäne. In der Literatur werden mögliche Kreuzreaktionen zu den Coronaviren SARS-CoV-1, MERS-CoV, HCoV-HKU1, HCoV-OC43, HCoV-NL63, HCoV-229E sowie bei Katzen und Schweinen vorkommende Coronaviren genannt.[155][160] Bei der Validierung der entwickelten Methoden zum Antikörpernachweis sind die diagnostische Sensitivität (Richtig-positiv-Rate) und Spezifität (Richtig-negativ-Rate) wichtige Qualitätskriterien, die mit Hilfe folgender Tabelle veranschaulicht werden.[154][161]
| krank (infiziert) | gesund (nicht infiziert) | |
|---|---|---|
| Test positiv | positiv (P) | falsch positiv (FP) |
| Test negativ | falsch negativ (FN) | negativ (N) |
Die Sensitivität des Testverfahrens gibt den Anteil der positiv Getesteten zur Gesamtheit der tatsächlich Infizierten an, die Spezifität hingegen gibt den Anteil der negativ Getesteten zur Gesamtheit der tatsächlich Nicht-Infizierten an, als Formeln ausgedrückt:
- $ {\text{Sensitivität}}={\frac {\text{P}}{{\text{P}}+{\text{FN}}}} $ bzw. $ {\text{Spezifität}}={\frac {\text{N}}{{\text{N}}+{\text{FP}}}} $
Für ein zuverlässiges Testergebnis werden für beide Kriterien Werte nahe 100 % angestrebt, eine hohe Sensitivität stellt sicher, dass kein Infizierter versehentlich übersehen wird, eine hohe Spezifität spricht dafür, dass kein „Fehlalarm“ (z. B. durch Kreuzreaktivität) ausgelöst wird.[161] Für die Methodenvalidierung serologischer Nachweise wird als Referenzmethode (auch als Goldstandard bezeichnet) der Neutralisationstest (NT),[161] im Detail der Plaque-Reduktions-Neutralisationstest (PRNT) verwendet. Für die als Proben eingesetzten Patientenseren gilt, dass bei den Patienten zuvor das SARS-CoV-2 durch PCR nachgewiesen wurde bzw. bei den Kontrollen die anderen Coronaviren oder weiteren Viren.[160] Für die Aussagekraft der Spezifität ist weiterhin wichtig, dass Proben mit Antikörpern gegen viele verschiedene Viren getestet werden.

In einem chinesischen Forschungslabor wurden im Januar 2020 erste ELISA-Tests durchgeführt, als Antigen wurde das Nukleokapsidprotein (N) eines Fledermaus-Coronavirus mit Ähnlichkeit zu SARS-CoV-2 verwendet. Damit ließen sich in Serumproben eines Patienten die Antikörper IgG und IgM nachweisen und deren Titer über mehrere Tage während des Krankheitsverlaufes bestimmen. In einem zweiten Test wurden Serumproben, die 20 Tage nach den ersten Symptomen entnommen wurden, untersucht. Alle Patientenseren, aber nicht die Seren von Gesunden zeigten eine stark positive IgG-Reaktion, einige Patientenseren zeigten zusätzlich eine IgM-Reaktion, was auf eine aktuelle Immunantwort, also eine momentane Infektion hindeutet.[63]
Seit März 2020 sind kommerzielle Antikörpernachweistests verfügbar. Im Rahmen der Validierung wurden zwei ELISA-Tests überprüft, die auf einer IgA- und IgG-Reaktion auf das S1-Protein basieren, dabei wurde eine eher geringe Spezifität festgestellt, da es zur Kreuzreaktivität mit dem Humanen Coronavirus OC43 kam und damit zu falsch positiven Ergebnissen. In der Sensitivität schnitt das IgA-ELISA besser ab. Allerdings basiert die Validierungsstudie nur auf einer geringen Anzahl von Patienseren, einmal n=10 von drei COVID-19-Patienten, zum anderen n=31 von neun COVID-19-Patienten.[160]
Zellkultur
Die Vermehrung des Virus zu Forschungszwecken in einer Zellkultur ist unter anderem in China, Australien, Frankreich, Deutschland und den USA gelungen.[14][162][163][164][165] Die chinesischen Wissenschaftler verwenden hierbei Epithelzellen des menschlichen Atemtrakts, die das mehrschichtige mukoziliäre Epithelgewebe (Flimmerepithel) simulieren, ebenso werden die Zelllinien Vero E6 und Huh-7 (isoliert aus humanem Leberkarzinom) eingesetzt.[14][63]
Behandlung
Für die Krankheit COVID-19 gibt es bisher keine spezifische Behandlung, eine Therapie zielt darauf ab, die Symptome zu lindern. Es wird jedoch untersucht, ob bereits bekannte Virostatika auch bei einer Infektion mit SARS-CoV-2 wirksam sind.
Vorbeugung
Entwicklung von Impfstoffen
Bereits unmittelbar nach Veröffentlichung der RNA-Sequenz des Virus wurde in mehreren Laboren mit der Impfstoffentwicklung begonnen.[166][167] Die internationale Impfstoffinitiative CEPI (Coalition for Epidemic Preparedness Innovations) plante, bis Mitte Juni 2020 erste Tests mit bis dahin entwickelten Impfstoffen durchzuführen. Dafür erhielten mehrere potentiell geeignete Unternehmen finanzielle Unterstützungen.[168] In Deutschland betraf dies u. a. die Tübinger Biotechnologiefirma Curevac, die zusammen mit dem Paul-Ehrlich-Institut an der schnellen Impfstoffentwicklung arbeitete.[169][170] Das Robert Koch-Institut verwies darauf, dass derzeit klinische Studien mit Impfstoffen gegen MERS-CoV laufen würden.[19] Allerdings sind klinische Studien lediglich der erste Schritt, es würde bei erfolgreichem Studienverlauf voraussichtlich frühestens in mehreren Monaten ein Impfstoff zur Verfügung stehen, der allerdings in einer ersten Phase sicher nicht für die gesamte Bevölkerung bereitgestellt werden könnte.[171]
Impfung gegen andere Infektionen
Die Berliner Senatsgesundheitsverwaltung empfahl Ende Februar 2020 allen Menschen über 60 Jahre und chronisch Kranken, ihren Impfstatus zu überprüfen und gegebenenfalls die Impfung gegen Pneumokokken (Impfstoffe wie Pneumovax 23 waren jedoch im März 2020 nur noch eingeschränkt lieferbar[172]) und Keuchhusten (Pertussis) durchführen oder auffrischen zu lassen. Da Menschen über 60 Jahren und chronisch Kranke durch SARS-CoV besonders gefährdet sind, seien sie vorsorglich zu schützen.[173][174]
Hygienemaßnahmen
Epidemiologie
SARS-CoV-2 verursacht die neuartige Erkrankung namens COVID-19 (für englisch corona virus disease 2019), die im Dezember 2019 in der Millionenstadt Wuhan der chinesischen Provinz Hubei auffällig wurde, sich im Januar 2020 in der Volksrepublik China zur Epidemie entwickelte und sich schließlich weltweit als COVID-19-Pandemie ausbreitete. Um einer Ausbreitung in Staaten ohne leistungsfähige Gesundheitssysteme entgegenzuwirken, rief die Weltgesundheitsorganisation (WHO) am 30. Januar 2020 die internationale Gesundheitsnotlage aus.[175] Am 11. März 2020 stufte die WHO die bisherige Epidemie zu einer Pandemie hoch.[176]
Meldepflicht
In Deutschland ist laut Infektionsschutzgesetz (IfSG) das Auftreten einer bedrohlichen übertragbaren Erkrankung an das zuständige Gesundheitsamt zu melden (§ 6 Abs. 1 Nr. 5 IfSG). Entsprechendes gilt für den Nachweis eines gefährlichen Erregers nach den in § 7 Absatz 2 IfSG bestimmten Umständen. Mit Wirkung vom 1. Februar 2020 wurde durch die Coronavirus-Meldepflichtverordnung eine Ausdehnung der Meldepflicht beschlossen. Das deutsche Bundesgesundheitsministerium konnte sich dabei auf eine Ermächtigung zum Erlass von Verordnungen berufen (§ 15 Absatz 2 IfSG). Nach dieser Verordnung sind sowohl der Verdacht einer Erkrankung, die Erkrankung und der Tod als auch der laborchemische Nachweis einer akuten Infektion mit dem neuartigen Coronavirus meldepflichtig. Die Meldung des Verdachts hat nur zu erfolgen, wenn der Verdacht sowohl durch das klinische Bild als auch durch einen wahrscheinlichen epidemiologischen Zusammenhang begründet ist. Dabei ist die Empfehlung[177] zu berücksichtigen, die das Robert Koch-Institut veröffentlicht. Ausnahmsweise muss man bei diesem Virus auch melden, wenn sich der Verdacht nicht bestätigt. Diese Verordnung gilt bis zum 1. Februar 2021,vorbeiVorlage:Warnung sofern nicht mit Zustimmung des Bundesrates etwas anderes verordnet wird. Am 29. April 2020 beschloss das Bundeskabinett einen Formulierungsvorschlag, nach dem Labore auch negative Testergebnisse melden sollen. Auch der Behandlungszustand zur Krankheit COVID-19 (einschließlich der Genesung) und der Serostatus sollen nach dem Zweiten Gesetz zum Schutz der Bevölkerung bei einer epidemischen Lage von nationaler Tragweite durch Ärzte und Gesundheitsämter gemeldet werden.[178][179] Am 14. Mai 2020 nahm der Bundestag einen entsprechenden Gesetzentwurf der Koalition an.[180] Am 15. Mai 2020 stimmte der Bundesrat zu.[181][182]
In Österreich besteht ebenfalls Anzeigepflicht und zwar nach dem Epidemiegesetz[183] von 1950 zusammen mit einer Verordnung.[184] Die Pflicht zur Anzeige besteht für Verdachts-, Erkrankungs- und Todesfälle aufgrund dieses Virus. Zudem wurde auch die Absonderungsverordnung[185] um das neue Coronavirus erweitert.[186]
Auch in der Schweiz existiert eine Meldepflicht.[187] Diese folgt aus dem Epidemiengesetz[188] der Schweiz in Verbindung mit der Epidemienverordnung[189] und der Verordnung des EDI über die Meldung von Beobachtungen übertragbarer Krankheiten des Menschen[190]. Nach Anhang 1 der Verordnung des EDI müssen Ärzte einen klinischen Verdacht und die Veranlassung einer erregerspezifischen Labordiagnostik und den nötigen epidemiologischen Zusammenhang melden. Nach Anhang 3 der Verordnung des EDI müssen Labore einen positiven und negativen Befund (also Nachweis) melden. Das Bundesamt für Gesundheit hat hierzu Verdachts-, Beprobungs-, Meldekriterien veröffentlicht.[191]
Weblinks
- COVID-19 (Coronavirus SARS-CoV-2). Übersichtsseite des Robert Koch-Instituts (RKI). In: rki.de. Robert Koch-Institut, 19. März 2020 (wird laufend aktualisiert).
- Antworten auf häufig gestellte Fragen zum Coronavirus SARS-CoV-2. In: rki.de. Robert Koch-Institut, 18. März 2020 (wird laufend aktualisiert).
- Coronavirus-Pandemie in Deutschland: Herausforderungen und Interventionsmöglichkeiten. (PDF; 158 kB) Nationale Akademie der Wissenschaften Leopoldina, 21. März 2020.
- Antworten auf häufig gestellte Fragen zum neuartigen Coronavirus (SARS-CoV-2). In: infektionsschutz.de. Bundeszentrale für gesundheitliche Aufklärung, 28. Februar 2020 (wird wochentäglich aktualisiert).
- Kann das neuartige Coronavirus über Lebensmittel und Spielzeug übertragen werden? Aktualisierte Fragen und Antworten des BfR vom 13. März 2020. In: bfr.bund.de. Bundesinstitut für Risikobewertung, 13. März 2020.
- Aktuelle Informationen zum Coronavirus. In: bundesgesundheitsministerium.de. Bundesministerium für Gesundheit (Deutschland), 29. Februar 2020 (wird laufend aktualisiert).
- Informationen zum Coronavirus. In: sozialministerium.at. Bundesministerium für Soziales, Gesundheit, Pflege und Konsumentenschutz (Österreich), 6. März 2020 (wird laufend aktualisiert).
- Neuartiges Coronavirus (COVID-19). In: sozialministerium.at, Infektionskrankheiten A-Z. Bundesministerium für Soziales, Gesundheit, Pflege und Konsumentenschutz (Österreich), 6. März 2020 (wird laufend aktualisiert).
- Neues Coronavirus. In: bag.admin.ch. Bundesamt für Gesundheit (Schweiz), 13. März 2020 (wird laufend aktualisiert).
- Q & A on COVID-19. In: ecdc.europa.eu. Europäisches Zentrum für die Prävention und die Kontrolle von Krankheiten, 16. Februar 2020, abgerufen am 29. Februar 2020 (Lua-Fehler in Modul:Multilingual, Zeile 149: attempt to index field 'data' (a nil value)).
- Weltgesundheitsorganisation: Coronavirus disease (COVID-19) outbreak. In: who.int. (Lua-Fehler in Modul:Multilingual, Zeile 149: attempt to index field 'data' (a nil value), wird laufend aktualisiert).
- Terra X: Das neue Coronavirus wissenschaftlich geprüft. In: zdf.de. 30. Januar 2020.
- Harald Lesch: Corona: Was weiß die Wissenschaft? Beitrag im Wissenschaftsmagazin Leschs Kosmos. In: Website ZDF. 24. März 2020 (43 min, verfügbar bis 17. März 2025).
- The Novel Coronavirus Pneumonia Emergency Response Epidemiology Team: Vital Surveillances: The Epidemiological Characteristics of an Outbreak of 2019 Novel Coronavirus Diseases (COVID-19) – China, 2020. 21. Februar 2020, abgerufen am 1. März 2020 (Lua-Fehler in Modul:Multilingual, Zeile 149: attempt to index field 'data' (a nil value)).
- Größte offizielle Sammlung an Experten-Antworten für Risikogruppe In: Österreichische Online-Plattform für chronisch Kranke, 30. März 2020, abgerufen am 30. März 2020.
- Podcast mit Christian Drosten im NDR, oder die Skripte zur Sendung im PDF
- WDR: Corona – die wichtigsten Fakten. In: Quarks-Extra. 4. April 2020, abgerufen am 28. März 2020 (Die Dauer die das kontaminierte Aerosol schwebend in der Luft verbringt bevor es zu Boden sinkt wird hier mit maximal 10 Minuten angegeben, das stimmt nicht überein mit dem neusten wissenschaftlichen Stand wo längere Zeiten genannt werden, ansonsten ist der Beitrag aktuell, sehr informativ und gut aufbereitet um die Thematik auch Nicht-Medizinern nahe zu bringen).
- Zita Aretz, Victor Nicolaus: Wie das Coronavirus unsere Zellen infiziert. In: n-tv. 5. April 2020, abgerufen am 6. April 2020 (Der Titel bezieht sich explizit auf SARS-CoV-2. Mit zahlreichen Schemazeichnungen bebildert inklusive Hygienemaßnahmen zur Vermeidung einer Infektion und deren Wirkungsweise).
Einzelnachweise
- ↑ 1,0 1,1 1,2 1,3 1,4 1,5 1,6
- ↑ Zur Rechtschreibung: „A species name is written in italics with the first word beginning with a capital letter.“ Vgl. How to write virus and species names. In: ICTV. Abgerufen am 26. April 2020.
- ↑ 3,0 3,1 3,2 3,3 ICTV: ICTV Taxonomy history: Severe acute respiratory syndrome-related coronavirus, EC 51, Berlin, Juli 2019; Email ratification March 2020 (MSL #35)
- ↑ Alissa Eckert, MS, Dan Higgins, MAM: ID#: 23312. In: Centers for Disease Control and Prevention (Hrsg.): Public Health Image Library (PHIL). 2020, abgerufen am 26. Februar 2020.
- ↑ National Institute of Allergy and Infectious Diseases: Novel Coronavirus 2019. In: flickr. Abgerufen am 18. März 2020.
- ↑ 6,0 6,1 Zur Rechtschreibung: „A virus name should never be italicized, even when it includes the name of a host species or genus, and should be written in lower case. […] The first letters of words in a virus name, including the first word, should only begin with a capital when these words are proper nouns (including host genus names but not virus genus names) or start a sentence.“ Vgl. How to write virus and species names. In: ICTV. Abgerufen am 26. April 2020.
- ↑
- ↑ Zur Rechtschreibung: „Like a species name, a higher taxon name is written in italics and begins with a capital letter.“ Vgl. How to write virus and species names. In: ICTV. Abgerufen am 26. April 2020.
- ↑ Hinweise zu Erkennung, Diagnostik und Therapie von COVID-19 Patienten (PDF). Hrsg.: STAKOB Geschäftsstelle am Robert Koch-Institut, Stand: 17. April 2020, Abgerufen am 30. April 2020.
- ↑ 10,0 10,1 Novel Coronavirus (2019-nCoV). (PDF; 1,0 MB) Situation Report – 22. WHO, 11. Februar 2020, abgerufen am 13. Februar 2020.
- ↑ Pneumonia of unknown cause – China. Disease Outbreak News(DONs) –WHO, 5. Januar 2020, abgerufen am 29. April 2020.
- ↑ Stephanie Hegarty: The Chinese doctor who tried to warn others about coronavirus. BBC News, 6. Februar 2020, abgerufen am 6. Februar 2020 (Lua-Fehler in Modul:Multilingual, Zeile 149: attempt to index field 'data' (a nil value)).
- ↑ Deutsche Welle (www.dw.com): Whistleblower-Arzt aus Wuhan gestorben | DW | 07.02.2020. Abgerufen am 12. Mai 2020 (Lua-Fehler in Modul:Multilingual, Zeile 149: attempt to index field 'data' (a nil value)).
- ↑ 14,0 14,1 14,2 14,3
- ↑ Pneumonia of unknown cause – China. In: Website der WHO. 5. Januar 2020, abgerufen am 14. Januar 2020 (Lua-Fehler in Modul:Multilingual, Zeile 149: attempt to index field 'data' (a nil value)).
- ↑ New virus surging in Asia rattles scientists. In: nature.com. 20. Januar 2020, abgerufen am 28. Januar 2020 (Lua-Fehler in Modul:Multilingual, Zeile 149: attempt to index field 'data' (a nil value)).
- ↑ Wuhan seafood market may not be source of novel virus spreading globally. In: ScienceMag des AAAS. 26. Januar 2020, abgerufen am 23. April 2020 (Lua-Fehler in Modul:Multilingual, Zeile 149: attempt to index field 'data' (a nil value)).
- ↑ WHO Statement Regarding Cluster of Pneumonia Cases in Wuhan, China. WHO, 9. Januar 2020, abgerufen am 14. Januar 2020 (Lua-Fehler in Modul:Multilingual, Zeile 149: attempt to index field 'data' (a nil value)).
- ↑ 19,0 19,1 Antworten auf häufig gestellte Fragen zum Coronavirus SARS-CoV-2. In: Website des Robert Koch-Instituts. 18. März 2020, abgerufen am 19. März 2020.
- ↑ 20,0 20,1 20,2 F. Wu, S. Zhao, B. Yu, Y.-M. Chen, W. Wang, Y. Hu, Z.-G. Song, Z.-W. Tao, J.-H. Tian, Y.-Y. Pei, M. L. Yuan, Y.-L. Zhang, F.-H. Dai, Y. Liu, Q.-M. Wang, J.-J. Zheng, L. Xu, E. C. Holmes, Y.-Z. Zhang: Wuhan seafood market pneumonia virus isolate Wuhan-Hu-1, complete genome. In: Website Nucleotide des National Center for Biotechnology Information (NCBI). Abgerufen am 4. Februar 2020.
- ↑ WHO: Laboratory testing for 2019 novel coronavirus (2019-nCoV) in suspected human cases. Auf: who.int vom 14. Januar 2020.
- ↑ Karola Neubert: Erster Test für das neuartige Coronavirus in China ist entwickelt. In: Website Informationsdienst Wissenschaft (idw). 16. Januar 2020, abgerufen am 9. Februar 2020.
- ↑ 23,0 23,1 23,2 23,3 Victor M. Corman, Tobias Bleicker, Sebastian Brünink, Christian Drosten, Olfert Landt, Marion P. G. Koopmans, Maria Zambon, Malik Peiris: Diagnostic detection of 2019-nCoV by real-time RT-PCR (protocol-v2-1). Hrsg.: Charité Virologie, Berlin. 17. Januar 2019, S. 1–13 (PDF, 1, MB [abgerufen am 1. Februar 2020]).
- ↑ 24,0 24,1 Unlocking the Genetic Code of the Novel Coronavirus: How COVID-19 Made the Leap From Animals to Humans, auf SciTechDaily vom 26. März 2020, Quelle: University of Sydney
- ↑ 25,00 25,01 25,02 25,03 25,04 25,05 25,06 25,07 25,08 25,09 25,10 25,11 Alexandre Hassanin: Coronavirus Could Be a ‘Chimera’ of Two Different Viruses, Genome Analysis Suggests, auf: sciencealert vom 24. März 2020 (Quelle: The Conversation)
- ↑ Verordnung über die Ausdehnung der Meldepflicht nach § 6 Absatz 1 Satz 1 Nummer 1 und § 7 Absatz 1 Satz 1 des Infektionsschutzgesetzes auf Infektionen mit dem erstmals im Dezember 2019 in Wuhan/Volksrepublik China aufgetretenen neuartigen Coronavirus („2019-nCoV“). CoronaVMeldeV. Bundesministerium der Justiz und für Verbraucherschutz, Bundesamt für Justiz (Deutschland), 30. Januar 2020, abgerufen am 4. März 2020.
- ↑ Neues Coronavirus. In: bag.admin.ch. Bundesamt für Gesundheit BAG, 13. März 2020, abgerufen am 15. März 2020 (Lua-Fehler in Modul:Multilingual, Zeile 149: attempt to index field 'data' (a nil value)).
- ↑ Fast 100 weitere Tote durch Covid-19 in China. Süddeutsche Zeitung, 12. Februar 2020, abgerufen am 13. Februar 2020 (Direkt aus dem dpa-Newskanal).
- ↑ Novel Coronavirus (2019-nCoV). (Memento vom 28. Januar 2020 im Internet Archive)
- ↑ Taxonomy ID: 2697049 Wuhan seafood market pneumonia virus. (Memento vom 3. Februar 2020 im Internet Archive)
- ↑ Ching-Tse Cheng: WHO declines to name new pneumonia after 'China' or 'Wuhan'. Taiwan News, 14. Januar 2020, abgerufen am 14. Januar 2020 (Lua-Fehler in Modul:Multilingual, Zeile 149: attempt to index field 'data' (a nil value)).
- ↑ WHO issues best practices for naming new human infectious diseases. Weltgesundheitsorganisation, 8. Mai 2015, abgerufen am 6. Februar 2020 (Lua-Fehler in Modul:Multilingual, Zeile 149: attempt to index field 'data' (a nil value)).
- ↑ Taxonomy Browser: Severe acute respiratory syndrome coronavirus 2, Taxonomy ID: 2697049. In: Website National Center for Biotechnology Information (NCBI). Abgerufen am 16. Februar 2020 (Lua-Fehler in Modul:Multilingual, Zeile 149: attempt to index field 'data' (a nil value)).
- ↑ 34,0 34,1 Lars Fischer, Alina Schadwinkel: Verursacht das Coronavirus Engpässe bei Medikamenten? Stammt das Virus aus dem Pangolin? Website Spektrum.de, 10. Februar 2020, abgerufen am 15. Februar 2020.
- ↑
- ↑ 36,0 36,1 36,2 36,3 Kristian G. Andersen, Andrew Rambaut, W. Ian Lipkin, Edward C. Holmes, Robert F. Garry: The Proximal Origin of SARS-CoV-2. In: virological.org, Quelle: ARTIC Network, 17. Februar 2020, Nature
- ↑ 37,0 37,1 Ben Hu, Lei-Ping Zeng, Xing-Lou Yang, Xing-Yi Ge, Wei Zhang et al.: Discovery of a rich gene pool of bat SARS-related coronaviruses provides new insights into the origin of SARS coronavirus. In: PLOS Pathogens, 30. November 2017, doi:10.1371/journal.ppat.1006698
- ↑ ICTV Master Species List 2018b.v2. MSL #34, März 2019
- ↑ 39,0 39,1 39,2 Xingguang Li, Junjie Zai, Qiang Zhao, Qing Nie, Yi Li, Brian T. Foley, Antoine Chaillon: Evolutionary history, potential intermediate animal host, and cross‐species analyses of SARS‐CoV‐2, in: Journal of Medical Virology, 27. Februar 2020, doi:10.1002/jmv.25731, PDF, PMID 32104911, reseachGate
- ↑ Xiaolu Tang, Changcheng Wu, Xiang Li, Yuhe Song, Xinmin Yao, Xinkai Wu, Yuange Duan, Hong Zhang, Yirong Wang, Zhaohui Qian, Jie Cui, Jian Lu: On the origin and continuing evolution of SARS-CoV-2, auf: National Science Review (NSR, Oxford Academic) vom 3. März 2020, nwaa036, doi:10.1093/nsr/nwaa036
- ↑ Wissenschaftler entdecken zwei Hauptuntertypen des neuartigen Coronavirus, auf: People's Dayly Online, China (deutsch)
- ↑ Jessica Hamzelou: Coronavirus: Are there two strains and is one more deadly?, auf NewScientist vom 5. März 2020 (mit Coronavirus ist hier ausschließlich SARS-CoV-2 gemeint – freier Artikel)
- ↑ SARS-CoV-2: Gibt es 2 unterschiedliche Virusstämme?, auf: aerzteblatt.de vom 9. März 2020
- ↑ Gibt es zwei Stämme von Sars-CoV-2?, auf: n-tv.de vom 10. März 2020
- ↑ Korinna Hennig, Christian Drosten: Coronavirus-Update: "Viren mutieren immer", Interview auf ndr.de vom 6. März 2020
- ↑ Peter Forster, Lucy Forster, Colin Renfrew, Michael Forster: Phylogenetic network analysis of SARS-CoV-2 genomes, auf: PNAS vom 8. April 2020, doi:10.1073/pnas.2004999117
- ↑ Genetic Study Identifies Three Variants of SARS-CoV-2 Coronavirus, auf: Sci-News vom 9. April 2020
- ↑ Kai Kupferschmidt: Der Stammbaum der Pandemie, auf: Spektrum.de vom 18. März 2020 (mit Bezug auf Christian Drosten und Andrew Rambaut)
- ↑ Amanda Woods: Iceland scientists found 40 mutations of the coronavirus, report says, auf: New Yor Post vom 24. März 2020
- ↑ Bo Elkjær: Forskere har sporet 40 mutationer af coronavirus – alene på Island, auf: www.information.dk (Iceland outlet Information) vom 24. März 2020, dänisch
- ↑ Vanessa Chalmers: Scientists in Iceland claim they have found FORTY mutations of the coronavirus – and admit seven cases can be traced back to 'a football match in England', auf: www.dailymail.co.uk (MailOnline) vom 24. März 2020
- ↑ Alexander Elliott: Two types of COVID-19 in one individual, auf: www.ruv.is (RÚV news), Island, vom 24. März 2020
- ↑ Poppy Askham: Patient Infected With Two Strains of COVID-19 In Iceland, auf: The Reykjavík Grapevine vom 24. März 2020
- ↑ 54,0 54,1 54,2 Lucy van Dorp, Mislav Acmana, Damien Richard, Liam P. Shawd, Charlotte E. Ford, Louise Ormond, Christopher J. Owen, Juanita Pang, Cedric C. S. Tan, Florencia A. T. Boshier, Arturo Torres Ortiz, François Ballou: Emergence of genomic diversity and recurrent mutations in SARS-CoV-2, in: In Press, Journal Pre-proof, doi:10.1016/j.meegid.2020.104351
- ↑ SARS-CoV-2: Mutationen könnten Ansteckungsfähigkeit weiter erhöhen, aerzteblatt.de vom 6. Mai 2020
- ↑ Nadja Podbregar: Wie stark ist das Coronavirus mutiert?, auf scinexx.de vom 8. Mai 2020
- ↑ Bette Korber, David D. Montefiori u. a., Spike mutation pipeline reveals the emergence of a more transmissible form of SARS-CoV-2, bioarxiv 30. April 2020
- ↑ 58,0 58,1 58,2 58,3 58,4 NCBI Database Nucleotide, txid2697049 (Severe acute respiratory syndrome coronavirus 2). In: Website Nucleotide des National Center for Biotechnology Information (NCBI). Abgerufen am 16. Februar 2020.
- ↑
- ↑ Wuhan seafood market pneumonia virus. In: Website Genome des National Center for Biotechnology Information (NCBI). Abgerufen am 16. Februar 2020.
- ↑ 61,0 61,1 61,2 61,3 61,4
- ↑ 62,0 62,1 Hufeisennasenfledermäuse. Schutzgemeinschaft Deutscher Wald, Oberursel vom 16. Dezember 2015
- ↑ 63,0 63,1 63,2
- ↑
- ↑ 65,0 65,1 65,2 65,3
- ↑ Genom-Analysen klären Herkunft von 2019-nCoV. In: Website Deutsches Ärzteblatt. 30. Januar 2020, abgerufen am 9. Februar 2020.
- ↑ 2019-nCoV: Erste Bilder vom Virus und Erkenntnisse zum klinischen Verlauf. In: Website Deutsches Ärzteblatt. 27. Januar 2020, abgerufen am 11. Februar 2020.
- ↑ https://link.springer.com/content/pdf/10.1007%2F978-3-8274-2241-5_14.pdf
- ↑ Qiu, Y.; Zhao, Y.; Wang, Q.; Li, J.; ZHou, Z.; Liao, C.; Ge, X. Predicting the Angiotensin Converting Enzyme 2 (ACE2) Utilizing Capability as the Receptor of SARS-CoV-2. Preprints 2020, 2020030091 (doi: 10.20944/preprints202003.0091.v1)
- ↑ Christian JA Sigrist, Alan Bridge, Philippe Le Mercier: A potential role for integrins in host cell entry by SARS-CoV-2. In: Antiviral Research. Band 177, Mai 2020, S. 104759, doi:10.1016/j.antiviral.2020.104759 (elsevier.com [abgerufen am 30. April 2020]).
- ↑ Hoffmann et al., SARS-CoV-2 Cell Entry Depends on ACE2 and TMPRSS2 and Is Blocked by a ClinicallyProven Protease Inhibitor, Cell (2020), https://doi.org/10.1016/j.cell.2020.02.052
- ↑ Blake Oberfeld, Aditya Achanta, Kendall Carpenter, Pamela Chen, Nicole M. Gilette: SnapShot: COVID-19. In: Cell. Band 181, Nr. 4, Mai 2020, S. 954–954.e1, doi:10.1016/j.cell.2020.04.013, PMC 7190493 (freier Volltext) – (elsevier.com [abgerufen am 15. Mai 2020]).
- ↑ Adedeji, Adeyemi & Severson, William & Jonsson, Colleen & Singh, Kamalendra & Weiss, Susan & Sarafianos, Stefan. (2013). Novel Inhibitors of SARS-CoV Entry acting by Three Distinct Mechanisms.. Journal of virology. 87. 10.1128/JVI.00998-13.
- ↑ RNA-Viren. In: www.spektrum.de.
- ↑ http://hdl.handle.net/2268/4138
- ↑ 76,0 76,1
- ↑
- ↑ Researchers trace coronavirus outbreak in China to snakes. In: Website EurekAlert! 22. Januar 2020, abgerufen am 26. Januar 2020 (Lua-Fehler in Modul:Multilingual, Zeile 149: attempt to index field 'data' (a nil value)).
- ↑ Coronavirus: How worried should we be? BBC News, 27. Januar 2020, abgerufen am 27. Januar 2020 (Lua-Fehler in Modul:Multilingual, Zeile 149: attempt to index field 'data' (a nil value)).
- ↑ 80,0 80,1 80,2 Chengxin Zhang et al.: Protein Structure and Sequence Reanalysis of 2019-nCoV Genome Refutes Snakes as Its Intermediate Host and the Unique Similarity between Its Spike Protein Insertions and HIV-1, in: American Chemical Society: J. Proteome Res. Vom 22. März 2020, doi:10.1021/acs.jproteome.0c00129; PrePrint, PrePrint Volltext (PDF) vom 8. Februar 2020
- ↑ NCBI: Bat coronavirus RaTG13 (no rank)
- ↑
- ↑ Mike McRae: Coronaviruses Similar to The COVID-19 One Have Just Been Found in Pangolins, auf sciencealert vom 27. März 2020 (mit „COVID-19“ ist nicht die menschliche Krankheit, sondern es sind allgemein Sarbecoviren gemeint, mit „Coronaviruses“ speziell nur SARS-CoV-2). Die Schuppentiere bzw. Teile derselben waren teilweise vom chinesischen Zoll beschlagnahmt worden.
- ↑ Tommy Tsan-Yuk Lam et al.: Identifying SARS-CoV-2 related coronaviruses in Malayan pangolins, in: Nature vom 26. März 2020, doi:10.1038/s41586-020-2169-0 (Preprint)
- ↑ Pangolins, Not Snakes, May Be Missing Link in Coronavirus Jump From Bats to Humans, auf: SciTechDaily vom 27. März 2020, Quelle: American Chemical Society
- ↑ Tina Hesman Saey: https://www.sciencenews.org/article/coronavirus-covid-19-not-human-made-lab-genetic-analysis-nature, auf ScienceNews vom 26. März 2020
- ↑ Rachel L. Graham, Ralph S. Baric: Recombination, Reservoirs, and the Modular Spike: Mechanisms of Coronavirus Cross-Species Transmission, in: ASM: Journal of Virology 84 (7), März 2010, S. 3134-3146, doi:10.1128/JVI.01394-09, PDF
- ↑ Marderhunde als Zwischenwirt? Drosten bringt neue Virusquelle ins Spiel, auf n-tv.de vom 26. April 2020
- ↑ Der Marderhund als Coronavirus-Schleuder?
- ↑ James D. Cherry, Paul Krogstad: SARS: The First Pandemic of the 21st Century
- ↑ Q&A on coronaviruses (COVID-19). Abgerufen am 6. März 2020 (Lua-Fehler in Modul:Multilingual, Zeile 149: attempt to index field 'data' (a nil value)).
- ↑ 92,0 92,1 Coronavirus: Nein, Hunde erkranken nicht an Covid-19. In: Der Spiegel – Wissenschaft. Abgerufen am 6. März 2020.
- ↑ Coronaviren können für Haustiere gefährlich werden. In: Frankfurter Neue Presse. 6. März 2020, abgerufen am 6. März 2020.
- ↑ Immediate notification. Tai Hang, Islands District, Hong Kong. Auf: oie.int. Weltorganisation für Tiergesundheit (OIE), 29. Februar 2020.
- ↑ Noah Higgins-Dunn: A dog in Hong Kong tests positive for the coronavirus, WHO officials confirm. In: CNBC. 28. Februar 2020, abgerufen am 29. Februar 2020 (Lua-Fehler in Modul:Multilingual, Zeile 149: attempt to index field 'data' (a nil value)).
- ↑ Weltorganisation für Tiergesundheit (OIE): Immediate notification. Tai Hang, Islands District, Hong Kong. Auf: oie.int vom 29. Februar 2020.
- ↑ Follow-up report No. 3 (Final report): COVID-19 (SARS-COV-2), Hong Kong. Likely human to animal transmission. Auf: oie.int vom 28. März 2020.
- ↑ Immediate notification. Pok Fu Lam, Southern District, Hong Kong. Auf: oie.int. Weltorganisation für Tiergesundheit (OIE), 21. März 2020.
- ↑ Fall in Belgien - Katze positiv auf Coronavirus getestet, auf n-tv.de vom 27. März 2020.
- ↑ Coronavirus: Belgian cat infected by owner. Auf: brusselstimes.com vom 27. März 2010.
- ↑ Bericht des Agriculture, Fisheries and Conservation Department, Hong Kong. Auf: oie.int. Weltorganisation für Tiergesundheit (OIE), 5. Mai 2020.
- ↑ Qiang Zhang et al.: SARS-CoV-2 neutralizing serum antibodies in cats: a serological investigation. Auf: bioRxiv, Preprint vom 3. April 2020, doi:10.1101/2020.04.01.021196.
- ↑ Coronavirus can infect cats — dogs not so much. Auf: nature.com vom 1. April 2020.
- ↑ Peter J. Halfmann et al.: Transmission of SARS-CoV-2 in Domestic Cats. In: The New England Journal of Medicine. Online-Publikation vom 13. Mai 2020, doi:10.1056/NEJMc2013400.
- ↑ Xuhua Xia: Extreme genomic CpG deficiency in SARS-CoV-2 and evasion of host antiviral defense, in: Molecular Biologa and Evolution, Academic Press, 14. April 2020, doi:10.1093/molbev/msaa094
- ↑ Evidence of Stray Dogs as Possible Origin of COVID-19 Pandemic, auf: SciTechDaily vom 14. April 2020, Quelle: University of Ottawa
- ↑ A Tiger In New York Has tested Positive For Coronavirus. Auf: sciencealert.com vom 6. April 2020. Quelle: Agence France-Presse.
- ↑ OIE: Immediate notification: SARS-CoV-2/COVID-19, United States of America. Bronx County, Bronx, New York. Auf: oie.int vom 6. April 2020.
- ↑ Young-Il Kim et al.: Infection and Rapid Transmission of SARS-CoV-2 in Ferrets. In: Cell Host & Microbe. Online-Vorabveröffentlichung vom 6. April 2020, doi:10.1016/j.chom.2020.03.023.
- ↑ Neues Coronavirus SARS-CoV-2: Flughunde und Frettchen sind empfänglich, Schweine und Hühner nicht. Auf: idw-online.de vom 2. April 2020.
- ↑ Jianzhong Shi et al.: Susceptibility of ferrets, cats, dogs, and other domesticated animals to SARS–coronavirus 2. In: Science. Online-Vorabveröffentlichung vom 8. April 2020, eabb7015, doi:10.1126/science.abb7015.
- ↑ Jon Cohen: From mice to monkeys, animals studied for coronavirus answers. In: Science. Band 368, Nr. 6488, 2020, S. 221–222, doi:10.1126/science.368.6488.221, Volltext
- ↑ Pelztiere erkranken massenhaft an Covid-19
- ↑ COVID-19: protect great apes during human pandemics. In: Nature. Band 579, 2020, S. 497, doi:10.1038/d41586-020-00859-y.
- ↑ NCBI: Human coronavirus OC43 (no rank)
- ↑ 116,0 116,1 Nadja Podbregar: Coronavirus: Sind auch Menschenaffen gefährdet? Auf: scinexx.de vom 30. März 2020.
- ↑ Elle Hunt: Orangutans and other great apes under threat from covid-19 pandemic, auf: NewScientist vom 2. April 2020
- ↑ Chuan Qin et al.: Reinfection could not occur in SARS-CoV-2 infected rhesus macaques. In: bioRxiv. 14. März 2020, Vorlage:BioRxiv, doi:10.1101/2020.03.13.990226.
- ↑ Keine Mehrfachansteckung mit SARS-CoV-2 bei Affen. (Nicht mehr online verfügbar.) In: Deutsches Ärzteblatt. Deutscher Ärzteverlag, 18. März 2020, archiviert vom Original am 19. März 2020; abgerufen am 19. März 2020.
- ↑ Können Menschen zweimal Covid-19 kriegen?, auf: n-tv.de vom 18. April 2019, Quelle: ntv.de, Amélie Bottollier-Depois, AFP
- ↑ Abishek Chandrashekar et al.: SARS-CoV-2 infection protects against rechallenge in rhesus macaques. In: Science. Online-Vorabveröffentlichung vom 20. Mai 2020, eabc4776, doi:10.1126/science.abc4776.
- ↑ Barry Rockx, Thijs Kuiken, Sander Herfst et al.: Comparative pathogenesis of COVID-19, MERS, and SARS in a nonhuman primate model. In: Science. Online-Vorabveröffentlichung vom 17. April 2020, abb7314, doi:10.1126/science.abb7314.
- ↑ Informationen der BAuA: Neuartiges Virus SARS-CoV-2 (bislang 2019-nCoV) durch den ABAS in Risikogruppe 3 eingestuft und Empfehlungen zur Labordiagnostik gegeben. In: Website der Bundesanstalt für Arbeitsschutz und Arbeitsmedizin (BAuA). 19. Februar 2020, abgerufen am 23. Februar 2020.
- ↑ 124,0 124,1 Beschluss 1/2020 des ABAS vom 19. 2. 2020 und Begründung zur vorläufigen Einstufung des Virus SARS-CoV-2 in Risikogruppe 3 und Empfehlungen zu nicht gezielten Tätigkeiten (Labordiagnostik) und gezielten Tätigkeiten mit SARS-CoV-2. (PDF; 140 kB) In: Website der Bundesanstalt für Arbeitsschutz und Arbeitsmedizin. 19. Februar 2020, abgerufen am 23. Februar 2020.
- ↑ Interim Laboratory Biosafety Guidelines for Handling and Processing Specimens Associated with Coronavirus Disease 2019 (COVID-19). In: Website der US-amerikanischen Centers for Disease Control and Prevention (CDC). 16. Februar 2020, abgerufen am 23. Februar 2020 (Lua-Fehler in Modul:Multilingual, Zeile 149: attempt to index field 'data' (a nil value)).
- ↑ 126,0 126,1 126,2 Victor M. Corman, Tobias Bleicker, Sebastian Brünink, Christian Drosten, Olfert Landt, Marion P. G. Koopmans, Maria Zambon, Malik Peiris: Diagnostic detection of 2019-nCoV by real-time RT-PCR (protocol-v2-1). Hrsg.: Charité Virologie, Berlin. 17. Januar 2019 (PDF, 1, MB [abgerufen am 10. April 2020]).
- ↑ 127,0 127,1 127,2 127,3 127,4 Victor M. Corman, Olfert Landt, Marco Kaiser, Richard Molenkamp, Adam Meijer, Daniel K. W. Chu, Tobias Bleicker, Sebastian Brünink, Julia Schneider, Marie Luisa Schmidt, Daphne G. J. C. Mulders, Bart L. Haagmans, Bas van der Veer, Sharon van den Brink, Lisa Wijsman, Gabriel Goderski, Jean-Louis Romette, Joanna Ellis, Maria Zambon, Malik Peiris, Herman Goossens, Chantal Reusken, Marion P. G. Koopmans, Christian Drosten: Detection of 2019 novel coronavirus (2019-nCoV) by real-time RT-PCR. In: Eurosurveillance. Band 25, Nr. 3, 23. Januar 2020, ISSN 1560-7917, S. 1–8, doi:10.2807/1560-7917.ES.2020.25.3.2000045.
- ↑ 128,0 128,1 128,2 128,3 128,4 Coronavirus outbreak: New test kit can detect the virus in just 15 minutes. In: Website MSN. 10. Februar 2020, abgerufen am 16. Februar 2020 (Lua-Fehler in Modul:Multilingual, Zeile 149: attempt to index field 'data' (a nil value)).
- ↑ 129,0 129,1 Victor M. Corman, Tobias Bleicker, Sebastian Brünink, Christian Drosten, Olfert Landt, Marion P. G. Koopmans, Maria Zambon, Malik Peiris: Diagnostic detection of 2019-nCoV by real-time RT-PCR (protocol-v2-1). Hrsg.: Charité Virologie, Berlin. 17. Januar 2019, S. 1 unten (PDF, 1, MB [abgerufen am 10. April 2020]).
- ↑ Produzent von Corona-Tests: Firma im Ausnahmezustand. In: Die Tageszeitung taz.de. 12. März 2020, abgerufen am 14. März 2020.
- ↑ Coronavirus disease (COVID-19) technical guidance: Laboratory testing for 2019-nCoV in humans. In: Website WHO. Weltgesundheitsorganisation (WHO), 2020, abgerufen am 15. März 2020 (Lua-Fehler in Modul:Multilingual, Zeile 149: attempt to index field 'data' (a nil value)).
- ↑ Stefan Nicola: A Berlin Biotech Company Got a Head Start on Coronavirus Tests. In: Bloomberg Businessweek. 12. März 2020, abgerufen am 14. März 2020 (Lua-Fehler in Modul:Multilingual, Zeile 149: attempt to index field 'data' (a nil value)).
- ↑ Frankfurter Forscher entwickeln schnelleren Coronavirus-Test. In: bz-berlin.de. 31. März 2020, abgerufen am 1. April 2020.
- ↑ Victor M. Corman, Tobias Bleicker, Sebastian Brünink, Christian Drosten, Olfert Landt, Marion P. G. Koopmans, Maria Zambon, Malik Peiris: Diagnostic detection of 2019-nCoV by real-time RT-PCR (protocol-v2-1). Hrsg.: Charité Virologie, Berlin. 17. Januar 2019, S. 3 (in Beschreibung von „Figure 2“) (PDF, 1, MB [abgerufen am 23. April 2020]).
- ↑ Victor M. Corman, Tobias Bleicker, Sebastian Brünink, Christian Drosten, Olfert Landt, Marion P. G. Koopmans, Maria Zambon, Malik Peiris: Diagnostic detection of 2019-nCoV by real-time RT-PCR (protocol-v2-1). Hrsg.: Charité Virologie, Berlin. 17. Januar 2019, S. 6, Titel 1, Absatz 3 (PDF, 1, MB [abgerufen am 23. April 2020]).
- ↑ Patrick DM, Petric M, Skowronski DM, Guasparini R, Booth TF, Krajden M, McGeer P, Bastien N, Gustafson L, Dubord J, Macdonald D, David ST, Srour LF, Parker R, Andonov A, Isaac-Renton J, Loewen N, McNabb G, McNabb A, Goh SH, Henwick S, Astell C, Guo JP, Drebot M, Tellier R, Plummer F, Brunham RC: An Outbreak of Human Coronavirus OC43 Infection and Serological Cross-reactivity with SARS Coronavirus. In: The Canadian journal of infectious diseases & medical microbiology = Journal canadien des maladies infectieuses et de la microbiologie medicale. November 2006, doi:10.1155/2006/152612, PMID 18382647, PMC 2095096 (freier Volltext) – (Can J Infect Dis Med Microbiol. 2006 Nov;17(6):330-6.).
- ↑ 137,0 137,1 Yafang Li, Lin Yao, Jiawei Li, Lei Chen, Yiyan Song, Zhifang Cai, Chunhua Yang: Stability issues of RT-PCR testing of SARS-CoV-2 for hospitalized patients clinically diagnosed with COVID-19. In: Journal of medical virology. 26. März 2020, doi:10.1002/jmv.25786, PMID 32219885 (Epub ahead of print).
- ↑ 138,0 138,1 Emily Feng, Amy Cheng: Mystery In Wuhan: Recovered Coronavirus Patients Test Negative ... Then Positive. In: NPR.org. National Public Radio, Inc. (USA), 27. März 2020, abgerufen am 14. April 2020 (Lua-Fehler in Modul:Multilingual, Zeile 149: attempt to index field 'data' (a nil value), teilweise irreführend: Der Satz: „There are false positives with these types of tests,“ bezieht sich nicht auf einen bestimmten Test, wie man vermuten könnte).
- ↑
- ↑ 140,0 140,1 140,2 140,3 140,4 Laboratory testing for 2019 novel coronavirus (2019-nCoV) in suspected human cases. In: Website WHO. Weltgesundheitsorganisation (WHO), 17. Januar 2020, abgerufen am 5. Februar 2020 (Lua-Fehler in Modul:Multilingual, Zeile 149: attempt to index field 'data' (a nil value)).
- ↑ Global Initiative on Sharing All Influenza Data (GISAID). In: Website der GISAID (Bundesrepublik Deutschland als Host (Datenbankanbieter)). Abgerufen am 5. Februar 2020 (Lua-Fehler in Modul:Multilingual, Zeile 149: attempt to index field 'data' (a nil value)).
- ↑ CDC Tests for 2019-nCoV. In: Website der US-amerikanischen Centers for Disease Control and Prevention (CDC). 5. Februar 2020, abgerufen am 10. Februar 2020 (Lua-Fehler in Modul:Multilingual, Zeile 149: attempt to index field 'data' (a nil value)).
- ↑ Stephanie Soucheray: CDC sends out nCoV test kits as Wisconsin confirms case. In: Website der University of Minnesota. CIDRAP – Center for Infectious Disease Research and Policy, 5. Februar 2020, abgerufen am 10. Februar 2020 (Lua-Fehler in Modul:Multilingual, Zeile 149: attempt to index field 'data' (a nil value)).
- ↑ Axel Schröder: Corona-Krise: So werden Virusnachweis-Kits produziert. In: Website Deutschlandfunk. 6. März 2020, abgerufen am 15. März 2020.
- ↑ RealStar SARS-CoV-2 RT-PCR Kit 1.0. (PDF; 596 kB) In: Website Altona Diagnostics. Februar 2020, abgerufen am 15. März 2020 (Lua-Fehler in Modul:Multilingual, Zeile 149: attempt to index field 'data' (a nil value)).
- ↑ 146,0 146,1 146,2 M. Parčina, U. V. Schneider, B. Visseaux, R. Jozić, I. Hannet, J. G. Lisby: Multicenter evaluation of the QIAstat Respiratory Panel – A new rapid highly multiplexed PCR based assay for diagnosis of acute respiratory tract infections. In: PLOS ONE. Band 15, Nr. 3, 12. März 2020, S. e0230183, doi:10.1371/journal.pone.0230183, PMID 32163484, PMC 7067435 (freier Volltext).
- ↑ New test kits for coronavirus approved in China. In: Website Xinhua. 30. Januar 2020, abgerufen am 10. Februar 2020 (Lua-Fehler in Modul:Multilingual, Zeile 149: attempt to index field 'data' (a nil value)).
- ↑ China's Jiangsu develops rapid test kit for coronavirus. In: Website Xinhua. 31. Januar 2020, abgerufen am 16. Februar 2020 (Lua-Fehler in Modul:Multilingual, Zeile 149: attempt to index field 'data' (a nil value)).
- ↑ Dean Koh: iHealthtech researchers working on Wuhan novel coronavirus (2019-nCoV) detection kit. In: Website mobi health news der National University of Singapore. 6. Februar 2020, abgerufen am 16. Februar 2020 (Lua-Fehler in Modul:Multilingual, Zeile 149: attempt to index field 'data' (a nil value)).
- ↑ M. Döhla, C. Boesecke, B. Schulte, C. Diegmann, E. Sib: Rapid point-of-care testing for SARS-CoV-2 in a community screening setting shows low sensitivity. In: Public Health. Band 182, 1. Mai 2020, ISSN 0033-3506, S. 170–172, doi:10.1016/j.puhe.2020.04.009, PMC 7165286 (freier Volltext) – (sciencedirect.com [abgerufen am 26. April 2020]).
- ↑ 151,0 151,1 151,2 Hildener Unternehmen entwickelt Schnelltest für Corona-Virus. In: wdr.de. 5. Februar 2020, abgerufen am 28. Februar 2020.
- ↑ 152,0 152,1 152,2 Pressemitteilung: QIAGEN announces worldwide shipments of QIAstat-Dx test kits for SARS-CoV-2. In: Website Qiagen. 26. Februar 2020, abgerufen am 28. Februar 2020 (Lua-Fehler in Modul:Multilingual, Zeile 149: attempt to index field 'data' (a nil value)).
- ↑ 153,0 153,1 Maike Telgheder: Diagnose Coronavirus: Die Industrie entwickelt Schnelltests. In: Website Handelsblatt. 26. Februar 2020, abgerufen am 28. Februar 2020.
- ↑ 154,0 154,1 154,2 154,3 154,4 154,5 Andreas Stiller: Ein tieferer Einblick in die Infektions-Tests gegen das Coronavirus SARS-CoV-2. In: heise online. 27. März 2020, abgerufen am 28. März 2020.
- ↑ 155,0 155,1 155,2 S. Y. Xiao, Y. Wu, H. Liu: Evolving status of the 2019 novel coronavirus infection: Proposal of conventional serologic assays for disease diagnosis and infection monitoring. In: Journal of Medical Virology. Band 92, Nummer 5, Mai 2020, vorab online veröffentlicht, S. 464–467, doi:10.1002/jmv.25702, PMID 32031264.
- ↑ 156,0 156,1 Juanjuan Zhao, Quan Yuan et al.: Antibody responses to SARS-CoV-2 in patients of novel coronavirus disease 2019. In: Clinical Infectious Diseases. 28. März 2020, doi:10.1093/cid/ciaa344.
- ↑ 2019-nCoV: Verdachtsabklärung und Maßnahmen – Orientierungshilfe für Ärztinnen und Ärzte. In: Website des Robert Koch-Instituts. 23. Januar 2020, abgerufen am 3. April 2020.
- ↑ Hinweise zur Testung von Patienten auf Infektion mit dem neuartigen Coronavirus SARS-CoV-2. In: Website des Robert Koch-Instituts (RKI). 20. Februar 2020, abgerufen am 25. Februar 2020.
- ↑ 159,0 159,1 LADR informiert Analytik Nr. 292 03/2020: Testung auf Antikörper gegen COVID-19 (SARS-CoV-2) verfügbar
- ↑ 160,0 160,1 160,2 160,3 Nisreen M.A. Okba1, Marcel A. Müller, Wentao Li, Chunyan Wang, Corine H. GeurtsvanKessel, Victor M. Corman, Mart M. Lamers, Reina S. Sikkema, Erwin de Bruin, Felicity D. Chandler, Yazdan Yazdanpanah, Quentin Le Hingrat, Diane Descamps, Nadhira Houhou-Fidouh, Chantal B.E.M. Reusken, Berend-Jan Bosch, Christian Drosten, Marion P.G. Koopmans, Bart L. Haagmans: Severe Acute Respiratory Syndrome Coronavirus 2−Specific Antibody Responses in Coronavirus Disease 2019 Patients. In: Emerging Infectious Diseases. Band 26, Nr. 7, doi:10.3201/eid2607.200841 (Juli 2020, vorab online veröffentlicht).
- ↑ 161,0 161,1 161,2 161,3 Antikörper und ihre Antigene; Methoden der mikrobiologischen Diagnostik. In: Helmut Hahn, Stefan H. E. Kaufmann, Thomas F. Schulz, Sebastian Suerbaum (Hrsg.): Medizinische Mikrobiologie und Infektiologie. 6. Auflage. Springer Verlag, Heidelberg 2009, ISBN 978-3-540-46359-7, S. 48–62, 140–150.
- ↑ Jef Akst: Australian Lab Cultures New Coronavirus as Infections Climb. In: Website The-Scientist. 29. Januar 2020, abgerufen am 4. Februar 2020 (Lua-Fehler in Modul:Multilingual, Zeile 149: attempt to index field 'data' (a nil value)).
- ↑ Institut Pasteur isolates strains of coronavirus 2019-nCoV detected in France. In: Website EurekAlert! 31. Januar 2020, abgerufen am 4. Februar 2020 (Lua-Fehler in Modul:Multilingual, Zeile 149: attempt to index field 'data' (a nil value)).
- ↑ Erstmals nCoV in Deutschland in Zellkultur isoliert. In: Website Institut für Mikrobiologie der Bundeswehr. 31. Januar 2020, abgerufen am 3. Februar 2020.
- ↑ CDC Grows 2019-nCoV Virus in Cell Culture. In: Website der US-amerikanischen Centers for Disease Control and Prevention (CDC). 6. Februar 2020, abgerufen am 10. Februar 2020 (Lua-Fehler in Modul:Multilingual, Zeile 149: attempt to index field 'data' (a nil value)).
- ↑
- ↑ Finn Mayer-Kuckuk: Suche nach Corona-Impfstoff: Das heiße Rennen. In: Die Tageszeitung: taz. 17. März 2020, ISSN 0931-9085 (taz.de [abgerufen am 18. März 2020]).
- ↑ Lars Fischer, Alina Schadwinkel: Verursacht das Coronavirus Engpässe bei Medikamenten? Stammt das Virus aus dem Pangolin? Website Spektrum.de, 10. Februar 2020, abgerufen am 15. Februar 2020.
- ↑ Sechste Infektion bestätigt: Kind in Bayern an Coronavirus erkrankt. In: Website tagesschau.de. 31. Januar 2020, abgerufen am 9. Februar 2020.
- ↑ Donald Trumps Test auf Coronavirus negativ ausgefallen. In: Der Spiegel – Politik. Abgerufen am 15. März 2020.
- ↑ Florian Schumann: Deutscher Coronavirus-Experte: „Wir müssen uns auf eine Pandemie einstellen“. In: Website Der Tagesspiegel. 13. Februar 2020, abgerufen am 15. Februar 2020.
- ↑ www.rki.de: Lieferengpässe.
- ↑ Gesundheitssenatorin begrüßt Ausweitung des Risikogebiets durch Robert-Koch-Institut. In: berlin.de. 26. Februar 2020, abgerufen am 29. Februar 2020.
- ↑ Coronavirus: Wann gilt der Katastrophenfall – und was folgt? In: rbb24.de. 26. Februar 2020, abgerufen am 29. Februar 2020.
- ↑ Weltgesundheitsorganisation: Statement on the second meeting of the International Health Regulations (2005) Emergency Committee regarding the outbreak of novel coronavirus (2019-nCoV). 30. Januar 2020, abgerufen am 30. Januar 2020 (Lua-Fehler in Modul:Multilingual, Zeile 149: attempt to index field 'data' (a nil value)).
- ↑ Tedros Adhanom Ghebreyesus: WHO Director-General's opening remarks at the media briefing on COVID-19 – 11 March 2020. In: Website WHO. 11. März 2020, abgerufen am 12. März 2020 (Lua-Fehler in Modul:Multilingual, Zeile 149: attempt to index field 'data' (a nil value)).
- ↑ Empfehlungen des Robert Koch-Instituts zur Meldung von Verdachtsfällen von COVID-19. Robert Koch-Institut, 24. März 2020, abgerufen am 26. März 2020.
- ↑ Gesetzentwurf der Fraktionen der CDU/CSU und SPD. Entwurf eines Zweiten Gesetzes zum Schutz der Bevölkerung bei einer epidemischen Lage von nationaler Tragweite. In: Bundestags-Drucksache 19/18967. 5. Mai 2020, abgerufen am 14. Mai 2020.
- ↑ Beschlussempfehlung und Bericht des Ausschusses für Gesundheit (14. Ausschuss). In: Bundestags-Drucksache 19/19216. 13. Mai 2020, abgerufen am 14. Mai 2020.
- ↑ Schutzpaket für mehr Coronatests und Pflege-Prämien beschlossen. Deutscher Bundestag, 14. Mai 2020, abgerufen am 14. Mai 2020.
- ↑ Ausgewählte Tagesordnungspunkte der 989. Sitzung am 15.05.2020. TOP 71 : Pandemieschutzgesetz II. In: Bundesrat Kompakt. Bundesrat, 15. Mai 2020, abgerufen am 15. Mai 2020.
- ↑ 246/20 Zweites Gesetz zum Schutz der Bevölkerung bei einer epidemischen Lage von nationaler Tragweite. Bundesrat, abgerufen am 15. Mai 2020.
- ↑ Epidemiegesetz 1950. Bundesrecht konsolidiert, Gesamte Rechtsvorschrift. In: RIS. 14. Juni 2018, abgerufen am 6. März 2020: „Der Anzeigepflicht unterliegen: […] (2) Der Bundesminister für Gesundheit und Frauen kann, […], durch Verordnung weitere übertragbare Krankheiten der Meldepflicht unterwerfen oder bestehende Meldepflichten erweitern.“
- ↑ 15. Verordnung des Bundesministers für Arbeit, Soziales, Gesundheit und Konsumentenschutz betreffend anzeigepflichtige übertragbare Krankheiten 2020. In: Bundesgesetzblatt für die Republik Österreich. 26. Januar 2020, abgerufen am 4. März 2020: „Der Anzeigepflicht nach dem Epidemiegesetz1950 unterliegen Verdachts-, Erkrankungs- und Todesfälle an 2019-nCoV („2019 neuartiges Coronavirus“).“
- ↑ Absonderungsverordnung. Bundesrecht konsolidiert: Gesamte Rechtsvorschrift für Absonderung Kranker, Krankheitsverdächtiger und Ansteckungsverdächtiger und Bezeichnung von Häusern und Wohnungen, Fassung vom 06.03.2020. In: RIS, Bundesrecht konsolidiert. 31. Januar 2020, abgerufen am 6. März 2020: „Bei Masern oder Infektion mit 2019-nCoV (“2019 neuartiges Coronavirus„) sind die Kranken und Krankheitsverdächtigen abzusondern oder nach den Umständen des Falles lediglich bestimmten Verkehrsbeschränkungen zu unterwerfen.“
- ↑ Verordnung vom 31. Jänner 2020. 21. Verordnung des Bundesministers für Soziales, Gesundheit, Pflege und Konsumentenschutz, mit der die Verordnung des Ministers des Innern im Einvernehmen mit dem Minister für Kultus und Unterricht vom 22. Februar 1915, betreffend die Absonderung Kranker, Krankheitsverdächtiger und Ansteckungsverdächtiger und die Bezeichnung von Häusern und Wohnungen geändert wird. In: Bundesgesetzblatt für die Republik Österreich. 31. Januar 2020, abgerufen am 6. März 2020: „In § 4 3. Satz wird nach dem Wort „Masern“ die Wortfolge „oder Infektion mit 2019-nCoV (“2019 neuartiges Coronavirus„)“ eingefügt.“
- ↑ Neues Coronavirus: Informationen für Gesundheitsfachpersonen. Verdachts- und Meldekriterien sowie Meldeformular. Bundesamt für Gesundheit, 5. März 2020, abgerufen am 5. März 2020.
- ↑ Bundesgesetz über die Bekämpfung übertragbarer Krankheiten des Menschen. Epidemiengesetz, EpG. Bundeskanzlei, 1. Januar 2017, abgerufen am 6. März 2020.
- ↑ Verordnung über die Bekämpfung übertragbarer Krankheiten des Menschen. (Epidemienverordnung, EpV) vom 29. April 2015 (Stand am 1. März 2019). Bundeskanzlei, abgerufen am 6. März 2020.
- ↑ Verordnung des EDI über die Meldung von Beobachtungen übertragbarer Krankheiten des Menschen. vom 1. Dezember 2015 (Stand am 1. Februar 2020). Bundeskanzlei, abgerufen am 6. März 2020.
- ↑ Meldeformulare. COVID-19 Meldung. Bundesamt für Gesundheit, 24. März 2020, abgerufen am 31. März 2020.








